编者按:
微生物组对人类健康的诸多方面都会产生影响,包括可能会影响癌症的进展及人体对癌症疗法的反应。近期的研究表明,微生物组对癌症的影响可能是直接的,也可能是间接的。
虽然,目前已有一些改变癌症患者微生物组组成的疗法在进行试验,但是关于微生物组与癌症及癌症疗法互作的机制我们尚不清楚。
由于癌症和微生物各自都是一个复杂的系统,所以要厘清两者之间的关系十分困难,那么,我们要如何探究微生物与癌症及癌症疗法之间的关系呢?
之前《热心肠日报》对近期发表于 Trends in Cancer 上的观点文章 The Cancer Microbiome:Distinguishing Direct and Indirect Effects Requires a Systemic View 作了简要解读,今天我们特别编译该文全文。希望该文能够为各位读者带来一些指导与启示。
以下是全文编译:
摘要
人体微生物组,指的是生活在人体内和人体表面的所有微生物,它可以影响癌症的发生、发展以及人体对癌症疗法(如癌症免疫疗法)的反应。搞清楚微生物组对癌症的影响机制可以提供新的诊断和治疗方法,尽管目前我们尚不清楚这些机制。
癌症相关的各个因素(如癌症本身的细胞-细胞互作、微生物、饮食、宿主因素和药物)的相互作用中存在复杂的反馈机制,任何一个单一的因素都不能解释整个癌症系统的所有表现。
所以,想要了解宿主相关微生物组在癌症系统中的作用,就需要一种多学科交叉的系统生物学方法,将微生物生态学、免疫学、癌细胞生物学和计算生物学结合起来。
微生物组与癌症:充满未知
一项经常被引用的统计数据显示,全球 20%的癌症是由传染性病原体引起的1。这一事实或许可以解释为什么癌症生物学倾向于将微生物(如 Box1 中的病毒和 Box2 中的细菌)视为要消除的致癌物。
构成人体微生物组的微生物影响着宿主生理的许多方面:营养摄取、药物代谢、炎症2,甚至行为3。它们很可能也会影响癌症的进展和治疗,并且这种影响可能有害,也可能有益。
了解微生物组如何直接或间接、有害或有益地影响癌症,可以为癌症预防、治疗和管理带来新的机会4。
尽管我们对微生物组有了越来越多的了解,但是许多知识鸿沟仍然存在:
微生物组是复杂的生态系统,具有时空动态性,这种动态性产生于微生物与宿主细胞之间的相互作用。而每种癌症本身也是一个生态系统,癌细胞会以错综复杂的动态方式相互作用,并与基质细胞相互作用。
常见因素如循环代谢物、全身免疫等,可同时影响并受到上述两种生态系统的影响。因此,微生物组与癌症之间的联系可能无法单纯地归为单一成分,要探索微生物组在癌症中的作用可能需要一种系统生物学方法。
2019 年,国际癌症微生物组联盟发表了一项声明:“目前没有直接证据表明人类共生微生物是癌症发病机制的关键决定因素”5。所以,区分微生物组对癌症的直接影响与间接影响至关重要。
当微生物直接接触癌变组织并影响其表现时,可能产生直接影响。当微生物组影响远端部位的肿瘤时,可能产生间接影响,例如肠道微生物组或可通过影响宿主生理或引发全身性炎症,从而影响其他部位的癌症进展(图1)。
尽管到目前为止微生物组直接影响癌症的例子很少,但过去十年的微生物组研究表明,微生物组能以多种方式影响宿主生理,其中的一些间接作用可能决定了癌症的发病机理。
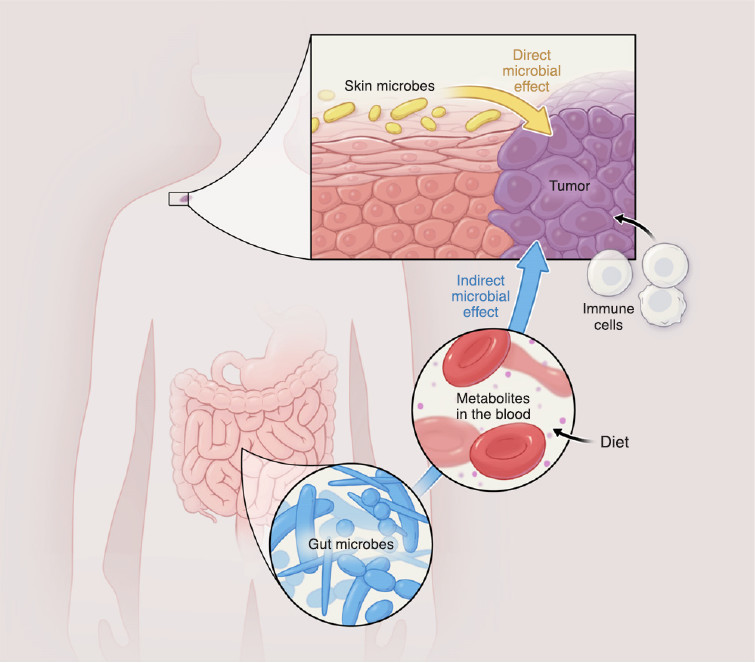
图1 人类微生物组可通过复杂的反馈回路与癌症相互作用,研究这种相互作用网络或需要系统生物学方法:微生物组对癌症的影响可能是直接的或间接的,这是一个重要的区别。
癌症组织中的微生物组可以与癌症发生直接相互作用,例如此处所示的假设情况:皮肤微生物组直接与黑素瘤相互作用。
存在于不同组织中的微生物组和癌症之间也可能发生间接相互作用,例如,肠道微生物组或可通过改变循环代谢物影响宿主生理,从而对皮肤癌的进展或宿主对治疗的反应产生间接影响。另外,饮食也许也能发挥作用,因为它也能影响代谢物的循环水平和微生物组组成。
Box1:致癌病毒和微生物组
Rous 于 1911 年发现病毒可以引起鸡的肉瘤,这是首次将微生物与癌症联系起来66。
Rous 从鸡的癌变组织提取出存在病毒的滤液,再把该滤液注射到健康的鸡体内,然后就引起了鸡的癌症。后来,Varmus、Bishop 及其同事对此背后的遗传机制开展了更深入的研究67。
癌症遗传学方面的第一个发现是关于病毒的,而这个发现开启了癌症分子生物学的时代。
病毒对我们将癌症理解为是一种遗传性疾病发挥了关键作用,而那些已经被发现可引起人类癌症的病毒则进一步证明了微生物与癌症之间的联系或可帮助癌症预防。
比如,针对乙型肝炎病毒和人乳头瘤病毒(HPV)的疫苗,成功地预防了这些病毒引起的癌症,如肝细胞癌和宫颈癌68。
虽然病毒与癌症之间有着联系,但不幸的是,微生物组研究中大多数都是关于细菌的,研究病毒的较少。
但是令人惊讶的是,微生物组的其他微生物也可能影响病毒引起的癌症的进展,这很重要,因为大多数感染致癌病毒的人往往不会发展成癌症,而决定病毒感染如何发展的因素仍不清楚。
一项针对波多黎各女性的研究表明,宫颈鳞状上皮内病变以及 HPV 感染会显著改变细菌和真菌的组成(图2)。因此,定植于宿主和环境交界处的宫颈阴道微生物组的变化,可能会影响对宫颈癌的易感性69。
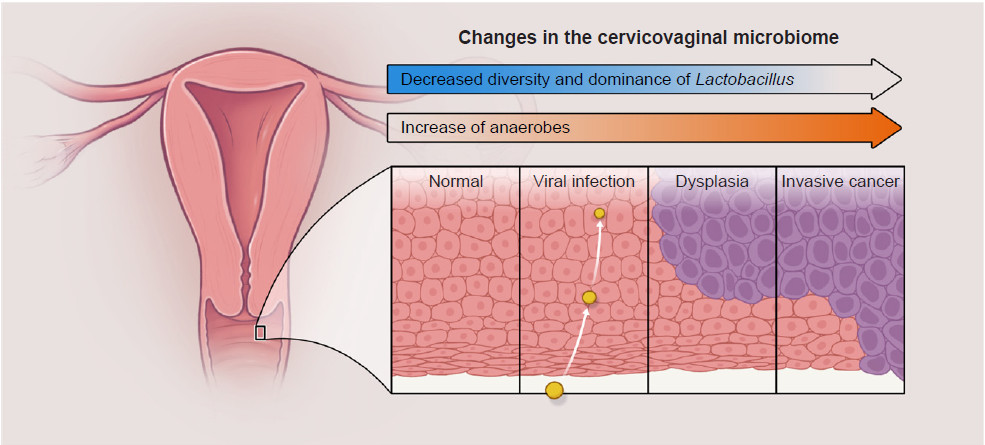
图 2 宫颈癌伴随着宫颈阴道微生物组组成的变化:微生物组从以乳酸杆菌为优势菌的群落变为富含严格厌氧菌(如 Sneathia sanguinigens 和 Gardnerella vaginallis)的群落。细菌性阴道炎与艾滋病病毒(HIV)和人乳头瘤病毒(HPV)的获得和传播,早产,盆腔炎和癌症的高风险相关。
病毒组研究出现了值得关注的挑战:宏基因组学和生物信息学流程如 ViromeScan70和virMine71可以找到已知的病毒,但想要发现未在数据库中列出的新病毒,则需要新的方法72。新的实验方法或许需要能从微生物组样本中选择性地检测出病毒,包括 RNA 病毒73。
解决这些技术挑战可以帮助我们更好地了解噬菌体在微生物组生态学中的作用。这在治疗上很重要:粪菌移植(FMT)和非生物性 FMT 的噬菌体移植与患者复发性艰难梭菌的阳性结果有关74~76,这一结果突出了微生物组内复杂的交互作用。
Box2:微生物在癌症中的双重作用
最著名的致癌细菌是幽门螺旋杆菌,全球 50%的人都携带这种细菌77,而且约 90%的胃癌都是由它导致的1。
20世纪上半叶,胃癌位列致命癌症名单之首。不过,或许由于卫生杀菌和抗生素的使用,胃癌的发病率在西方国家有所下降,因为一些用于治疗其他感染的抗生素可能无意中杀死了幽门螺旋杆菌78。
然而,幽门螺旋杆菌的例子说明了一个悖论:因为在它导致人类胃癌的同时,又被认为是胃微生物组的正常组成成分,可以在人与人之间传播,与人类共同进化79。
而且,幽门螺旋杆菌的定植甚至可能有益于人类健康,因为去除幽门螺旋杆菌会增加许多疾病的风险,比如严重的胃食管反流病及其后遗症、Barrett食管炎和食管腺癌80。它还可以预防哮喘、多发性硬化症和炎性肠病(IBD)81~83,尽管它也是 Ⅱ 型糖尿病和其他一些疾病的危险因素84,85。
因此,幽门螺旋杆菌可能既可以伤害人体又可以有益于人类健康。
这种双重性也适用于其他微生物86。或许,有害和有益之间的区别在于由微生物引起的炎症类型。
人体免疫系统是一种微妙的平衡,适度的免疫反应对于机体稳态至关重要:太弱的免疫反应可能无法抵御感染,但过度反应可能会破坏组织或引起自身免疫性疾病。
IBD 是一种慢性肠道炎症,可改变肠腔内微生物组的组成,即导致革兰氏阴性菌的富集。IBD 患者罹患结直肠癌的风险较高,这可能是由于肠腔内的一些细菌会促进癌症的发生,如一些 pks+ 大肠杆菌32。
然而,微生物引起的炎症也可以用来对抗癌症。如卡介苗(BCG),一种由牛分枝杆菌减毒菌株制备的疫苗,是治疗膀胱癌最成功的生物疗法之一87。
此外,微生物组对癌症的影响形式远远不止于存在或不存在某些特定微生物,即便出现某个单一的微生物对癌症具有影响,这很可能是例外,而非普遍规律。
想要了解癌症中的微生物组可能需要掌握生态网络的相关知识,以区分稳态网络和致癌网络,确定这些网络是静止的还是可塑的,并设计将致癌网络转换回稳态网络的方法。
微生物组通过代谢对癌症的间接影响
宿主生理活动的一个重要方面是:营养物质和代谢物循环水平的系统平衡。而该平衡已经被证实会被肠道微生物组所影响6。
微生物组的一项开创性研究表明,把肥胖小鼠的肠道微生物移植到瘦的无菌小鼠中时,受体小鼠会变得肥胖2。重要的是,接受来自肥胖供体微生物的小鼠比接受来自非肥胖供体微生物的小鼠,获得更多的脂肪,即使两组消耗相同数量的卡路里。
饮食中或者由宿主释放的代谢物进入肠道后,会被肠道微生物催化的生物反应所转化。宿主可以吸收或重吸收一些微生物转化的代谢产物,于是这些代谢产物进入了循环系统7,可以到达远端组织并影响那里的癌症进展。
例如,在肝癌中,肠道共生微生物可以将一级结构的胆汁酸代谢为二级结构的胆汁酸,二级结构的胆汁酸可以再循环,并调节自然杀伤T细胞向肝癌细胞募集8。
肠道中的一些细菌可以代谢雌激素,有潜力改变绝经后雌激素受体阳性乳腺癌的风险,尽管乳房远离肠道9,10。
在男性肠道和尿道中,微生物可以利用宿主的糖皮质激素生成 11-氧雄激素,这可能会导致前列腺癌11~13。
抗生素可以破坏肠道微生物组并影响血浆中代谢物的水平14,这可能会影响远端组织中与癌症相互作用的宿主细胞。
使用抗生素会通过破坏微生物组间接影响的两种代谢物——三甲胺 N-氧化物和甜菜碱,这两种代谢物会对小鼠腹腔的巨噬细胞产生影响,改变其表型并影响动脉粥样硬化相关的标志物15。
在这个例子中,抗生素治疗影响微生物组,然后微生物组以损害宿主健康的方式影响循环代谢物的水平。
然而,调控微生物组可以改变巨噬细胞标记物这一点,或会启发产生新的抗癌策略。
因为巨噬细胞有多种多样的表型,其中的一些表型实际上可以促进肿瘤的生长16。设计一种微生物组,使某些代谢物的循环水平降低,从而调控巨噬细胞。
例如乳酸可以使肿瘤相关巨噬细胞极化,如果降低乳酸的循环水平17,18,可能可以减弱这些巨噬细胞的肿瘤促进表型。
肠道微生物组还可能影响中枢神经系统、情绪和行为,并对癌症产生影响。微生物组与动物行为之间的联系是一个热门话题19,但这种联系可能是古老的:动物一直与微生物共存,且与能够影响行为的共生微生物保持共生关系3。
大脑的奖励系统是情绪过程中的关键回路,可以增强抗肿瘤免疫反应。在最近一项小鼠的研究中,用化学遗传学手段激活大脑的奖励系统可以缩小肺部的肿瘤20。
这里面的机制涉及一个级联效应:奖励系统减弱了骨髓(主要免疫部位)去甲肾上腺素能的输入,导致髓源性抑制细胞的免疫抑制降低,从而引发抗肿瘤反应。
而微生物组则可以间接影响宿主的奖励系统21。具体来说,因为有些药物会影响奖励系统,而微生物组可能会影响宿主对这些药物的反应22。因此,微生物组原则上可以通过改变宿主的心理状态,来增强宿主的抗肿瘤免疫,并间接减缓癌症的进展。
基于这一原则的癌症疗法,在今天看来似乎是科幻的,但益生菌用于减轻疼痛的功能,即利用益生菌直接或间接增强止痛剂如阿片类药物的作用正在研究中(图2)23。
至少在没有其他治疗方法时,或许可以考虑利用微生物组和中枢神经系统之间的相互作用来控制癌症症状。
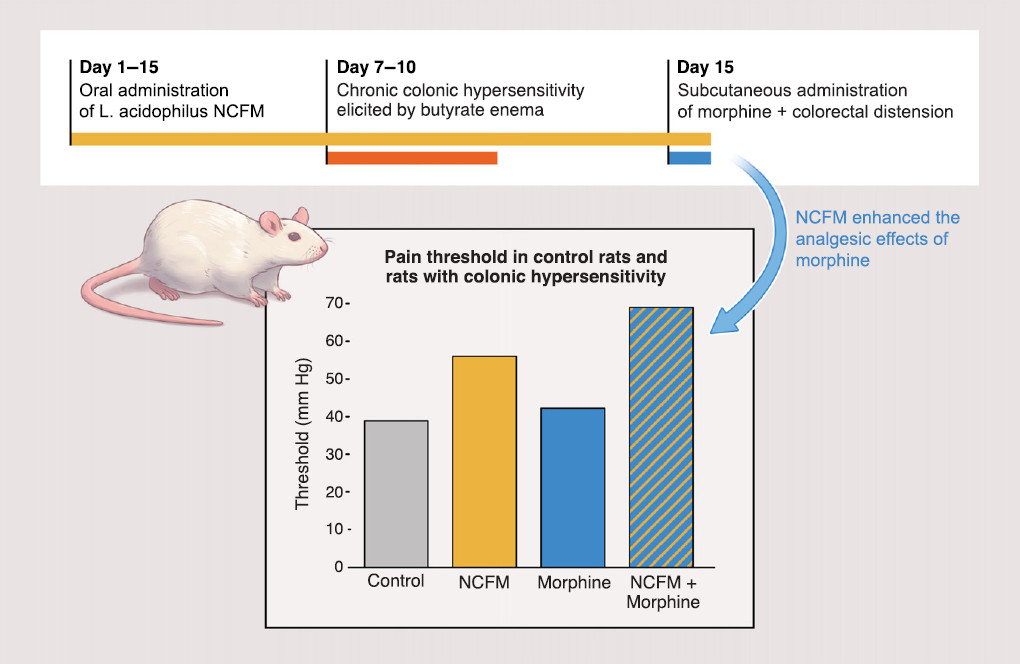
图 3 微生物组工程可以减轻癌症患者的疼痛:在小鼠模型中,与对照组相比,益生菌嗜酸乳杆菌 NCFM 菌株减轻了小鼠的疼痛,并增强了阿片类止痛剂的作用23。
想要充分利用微生物组对抗癌症的潜力,就需要更好地了解共生微生物是如何起作用的。
幸运的是,微生物组领域的研究正在迅速地从分类学研究扩展到功能研究(Box3)。
一些有益的功能可能归因于特定的微生物,例如双歧杆菌属的特定菌株能促进小鼠的抗肿瘤免疫24。
然而,其他功能,例如代谢能力,可能涉及到系统发育树上众多的微生物。假设同一进化枝内的微生物功能保守,那么可以用这几种微生物来发挥相同的功能25。
此外,关于微生物组组成的大人口样本的研究可以揭示共生微生物组的成员与癌症发病率之间的新联系(Box4)。
Box3:研究微生物组在癌症中作用的新兴技术
研究微生物组在癌症中的作用需要区分直接影响和间接影响,因为它们需要不同的方法,甚至不同的技术。
直接影响属于“肿瘤微生物组”的微生物,它们可以直接接触癌细胞并影响其表现。空间问题和肿瘤异质性起着重要作用。
这些研究可能需要复杂的原位成像技术,使空间结构保持完整。
我们可以利用多重杂交技术同时标记多个参与者:细菌、粘液,甚至肠细胞中基因的表达水平88,89。前景良好的杂交手段包括 CLASI-FISH,它使用细菌探针的组合来显示微生物的空间结构90,还有 MERFISH,它揭示了单细胞中的基因表达91。
但是,多重杂交手段会破坏样品,这意味着该技术不能用于跨时间跟踪同一的样品。
不过,这些方法确实提供了高分辨率的横断面数据,非常适合研究空间组织:这些数据可以产生新的基于智能体的计算模型,来描述癌细胞和微生物细胞在肿瘤中如何分布。
此类软件包如 PhysiCell,它结合了扩散梯度和机械过程的模拟92,可以扩展到细菌细胞,以用于研究介导癌细胞和微生物组互作的炎症反馈机制。
模型开发应该建立在建模者和实验者密切合作的基础上,以促进实验和模拟之间的反馈。
这些方法应将微生物组成和功能与其他相关数据整合,用于系统建模,如微生物的栖息地、宿主信息以及其它组学数据等。要分析微生物组的间接影响,就需要有一种系统性研究微生物组功能的方法。
一些生物信息学工具已经可用于研究微生物组功能,例如 BugBase93和 PICRUSt294,它们试图使用粗糙的表型类别(如“厌氧菌”,“葡萄糖利用菌”和“反硝化菌”)来量化共生微生物的功能组成。
其他工具,如 QIIME 295,努力为微生物组的多组学研究提供平台,整合不同类型的微生物组数据,如分类学数据、宏基因组和代谢组数据,以支持研究同时有哪些微生物存在、它们的潜在功能和代谢活动,能够超越简单的微生物组关联研究,来研究微生物组机制。
功能研究在应用微生物学的其他领域已经很常见,例如废水的生物处理96,并且功能研究可能是连接微生物组组成和功能的重要工具。
Box4:环境对微生物组成的影响及其在癌症发病中的作用
大样本的微生物组研究可以帮助我们了解癌症发病率的变化,并引出微生物与癌症背后的新机制。
工业化人群的微生物组,不同于保留前工业时代生活方式的人群,如哈扎人(坦桑尼亚的狩猎采集者)97,98。工业化人群的微生物组多样化变低了,这也许解释了:为什么在我们与微生物共生关系受到影响最大的一些地区,自身免疫和炎症性疾病的发病率也高99。
抗生素、饮食中的纤维量100、食品添加剂101,102,甚至过度卫生103也会改变微生物组的组成。所有这些变化都可能影响免疫反应,引起自身免疫问题,并减弱我们对抗病原体的能力99,所有这些都是癌症的危险因素。
最近一项关于美国移民人口微生物组的研究(图 4)显示,移民的肠道微生物组在来到美国后发生了变化:在迁移后,移民肠道中的原生菌株和功能,立即被美国人群的典型菌株和功能所取代104。
移民在美国停留的时间越长,他们微生物组的变化就越大。下一代的变化甚至更大,并且重要的是,这种变化与肥胖相关。
移民微生物组组成的变化可能仅仅是由于饮食的变化。然而,尚不清楚不同的肠道微生物是否可能来自其他人类,生水果或蔬菜,动物甚至其他环境来源。微生物多样性的丧失可能是由于不同饮食施加的选择压力,但也可能是由于美国环境中的微生物组不如原来的国家多样化。
尽管如此,这说明了迁移会对微生物组多样性产生影响,并且还会进一步导致癌症相关的健康问题(如肥胖症),这证明了有必要建立用于微生物组重建的微生物库。
非营利性 Microbiota Vault 拥有一个微生物库,名为“丢失的菌群”,以拯救现代社会耗尽的微生物多样性105。
FMT 可以成功治疗艰难梭菌结肠炎,所以另一家非营利性企业 Open Biome Project 保存了大量的 FMT 样本库76,106。
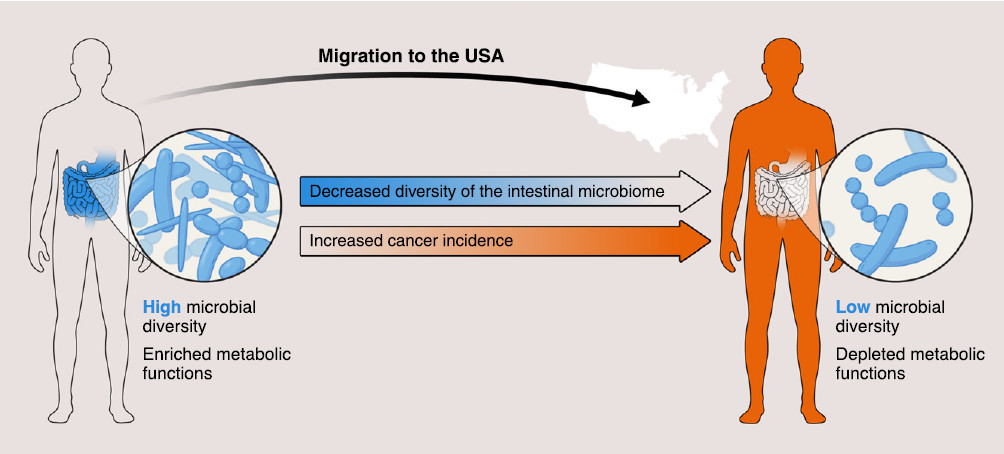
图 4 移民到美国会降低肠道微生物群的多样性:这可以解释为什么在美国移民的某些人群中,特定疾病(包括肥胖和某些癌症)的发病率增加。研究这种有趣的现象可以为微生物组在癌症中的作用提供新的线索。
微生物组对癌症的直接影响
关于微生物组对癌症直接影响的研究,大多数来自于自然定植在肿瘤组织中的共生微生物:结直肠癌的肠道微生物组,肺癌的肺微生物组,黑色素瘤的皮肤微生物组,宫颈癌和子宫内膜癌的阴道微生物组等。
但是,在一些出人意料的组织中的微生物,也可以影响癌症进展和对治疗的反应。胰腺癌可能会因医疗干预而意外引入细菌26~28,而结直肠癌会在转移时,把肠道细菌带到其他组织,如肝脏29。
无论这个组织是否已经被微生物定植,组织中的微生物和癌细胞之间的直接相互作用仍然知之甚少。
该研究领域将受益于体内模块化实验的系统,在该系统中,每个成分都可以独立地改变,从而揭示癌细胞和微生物之间相互作用的机制。
结直肠癌是美国死亡率第二高的癌症30。结直肠癌的关键风险因素是遗传易感性,但其他因素(如饮食、生活方式和微生物组组成)实际上占大多数31。
系统方法可能有助于理清这些风险。
结直肠癌已经有相应的动物模型,例如 Il10-/-小鼠,可以提供模块化实验系统。根据遗传背景,高达 100%的 Il10-/-小鼠会患有结肠炎;如果再用氧化偶氮甲烷(一种结肠特异性致癌物)处理,60~80%的结肠炎小鼠会出现结肠肿瘤32。
结直肠癌本身就是一种分阶段发展的模块化疾病(modular disease)33。微生物组直接定植于组织,随着疾病的进展,微生物组中的细菌、病毒和真菌的组成发生变化34~36。
完善的疾病模型与相应的手段的结合,可以让癌症微生物组研究辨别微生物在不同疾病阶段的作用和机制37。
西方饮食含有许多可以引发肠道肿瘤的营养素38。
这些饮食中含有丰富的动物蛋白质和脂肪,可以促进胆汁酸分泌到胃肠道中。胆汁酸会重吸收到宿主的循环系统中,肠道共生微生物可以在这之前将它们代谢。
一种名为 Bilophila wadsworthia 的微生物,可以将牛磺酸转化为乙酸盐和氨,并释放出致癌的硫化氢39。然而,即使特定微生物如 B.wadsworthia 会促进癌症的进展,其它微生物也可能对该过程产生影响,这意味着所涉及的机制必须要系统地探讨。
空间结构也可能起到关键作用。例如,生物膜结构与结直肠癌的发展位置有关40,并且在临床前模型中,生物膜会促进癌症的发生41。
转移使局部癌症转变为扩散性疾病,这是许多癌症从可治愈阶段演变为不可治愈阶段的关键转折点。
最近的一项研究表明,微生物可以与转移的癌细胞一起从原发肿瘤部位扩散到远端部位。
这项研究关注的是 Fusobacterium nucleatum,它是结直肠癌中最常见的细菌之一。患者活检报告表明,肠道微生物组中的 Fusobacterium 和其他细菌从原发肿瘤部位转移到远端部位(图 5)。
给小鼠异种移植原发性结直肠腺癌表明,癌细胞确实可以携带活的 Fusobacterium 并将其运输到转移部位。
在传代几次后,癌细胞依然可以保持这种携带细菌的能力,这种能力对于恶性肿瘤来说是很重要的。因为用抗生素治疗小鼠可以减少细菌量,并抑制肿瘤的生长。这些结果表明,抗菌药物干预或可帮助治疗与 Fusobacterium 相关的结直肠癌42。
然而,直接与癌细胞相互作用的细菌实际上可以引起局部炎症,来帮助治疗癌症43。这是细菌双重作用的额外证据。
当我们认为细菌可以导致耐药性时,这种相互作用就变得更加复杂。
一些细菌可以将化疗药物吉西他滨(2’,2’-二氟脱氧胞苷)代谢成其不活跃的形式,2’,2’-二氟脱氧尿苷。细菌要表达一种特定亚型的胞苷脱氨酶,才能使癌症药物失活,这种酶主要存在于 γ-变形菌中。
皮下肿瘤小鼠模型的实验表明,生活在肿瘤内的 γ-变形菌可以增加肿瘤对吉西他滨的耐药性。吉西他滨和环丙沙星(一种可以杀死 γ-变形菌的广谱抗生素)的联合疗法,阻止了对癌症药物的耐药性26,这是如何在癌症治疗中使用抗生素的另一个例子。

图 5 转移可以将原发性肿瘤微生物组的细菌携带到远端组织:最近的一项研究42调查了来自同一患者的结直肠癌和肝转移的活检样本。在原发性肿瘤中发现的细菌,包括 Fusobacterium nucleatum,也在转移部位被发现,表明它们与转移细胞一起扩散。小鼠实验验证了这些发现,并表明抗生素治疗可以减小肿瘤体积。
微生物组与癌症免疫疗法
想要了解微生物、免疫和癌症之间错综复杂的反馈,以及它们对免疫疗法的影响,可能需要多学科的方法和系统的视角。
免疫疗法可以治疗晚期黑色素瘤,也可能适用于其他癌症。使用检查点抑制剂,使免疫系统恢复功能,可以使高达 60%的转移性黑色素瘤患者得到长时间的缓解44。
最近发表的三篇论文表明,病人的肠道微生物组可以影响他们对免疫疗法的反应45~47。这些研究发现,对检查点抑制剂有反应的患者和没有反应的患者在肠道微生物组成上存在差异。
有趣的是,如果要确定哪些特定的微生物对免疫疗法的成功至关重要,这三项研究得到的答案并不一致48。
除了缺乏关于哪些共生微生物最重要的共识外,肠道微生物组影响免疫疗法的机制也尚不清楚49。
对检查点抑制剂有反应的患者的微生物组可以诱导 CD4 和 CD8 T 细胞产生干扰素(IFN)-γ 和颗粒酶 B,这将有助于招募抗肿瘤巨噬细胞。与肠道微生物组完整的小鼠相比,无菌小鼠和经过抗生素治疗的小鼠用检查点抑制剂治疗黑色素瘤的成功率较低24。
一些研究甚至认为,对检查点抑制剂有反应的患者中有特定细菌的富集,如 Bacteroidetes thetaiotaomicron 和 Faecalibacterium prausnitzii 50,这些微生物也许可助于使用精确的益生菌治疗,以提高检查点抑制剂的疗效。
最近的一项研究表明,肠道细菌产生的短链脂肪酸(SCFAs)促进了抗原激活的 CD8+ T 细胞的记忆潜能51。这一重要发现解决了不同团队提出的同一个疑问:分类学上不同的微生物对健康的影响是否相同。
有几个团队已经确定了 F.prausnitzii 存在于对检查点阻断有反应的患者中,并且 F. prausnitzii 是产生 SCFAs(包括丁酸酯)的关键梭菌。
关于 IBD 和肠道移植物抗宿主病的文献表明,这些梭菌属的 XIVa 簇和 IV 簇细菌产生 SCFAs 后会诱导调节性 T 细胞和白细胞介素(IL)-10,产生一个更耐受和抗炎的环境。
然而在癌症免疫疗法中,肠道微生物如 F. prusnitzii 是有益的,这看似矛盾,但最近的研究表明,这些 SCFA 可能可以根据不同的环境和宿主特异性因素而产生不同的表型51。
这一条件机制解释了产生 SCFA 的梭菌对免疫检查点阻断效果有影响的原因,且与先前提出的观点相呼应,即几种肠道微生物可能有一个共同的功能(例如产生 SCFA,丁酸),以及它们产生的代谢物发挥着重要的功能。
研究免疫疗法患者的肠道微生物组还有其他原因:该疗法有副作用,它本身可以影响肠道微生物组,引起更多并发症。
检查点抑制剂可引起免疫介导的结肠炎52。
一项前瞻性研究发现,接受伊匹单抗治疗的转移性黑色素瘤患者中,一些肠道菌群有前结肠炎特点的患者,最后往往会患有结肠炎53。这些患者拥有较少的拟杆菌门细菌,并且参与多胺转运和 B 族维生素生物合成的基因表达也较低。
如果这些生物标志物确实解释了为什么一些患者会患上结肠炎,那么这一发现可能会有助于干预措施改善微生物组,并降低癌症免疫疗法引起炎症并发症的风险。
更广泛地说,前瞻性研究表明,免疫疗法或可引起肠道炎症,然后改变微生物组的组成,激活可导致更多并发症的反馈回路。
值得注意的是,在一个病例报告中,连续的粪菌移植(FMT)减轻了患者因程序性细胞死亡蛋白 1(PD-1)治疗诱导的难治性结肠炎54。
治疗后恢复微生物组
放疗和化疗等癌症疗法会破坏微生物组并导致并发症。
患有前列腺癌、妇科恶性肿瘤或胃肠癌的患者,通常会接受盆腔放疗,一般都是非常有效的,比如该方法对 90%的前列腺癌起效。
然而,盆腔放疗会导致慢性腹泻,目前尚无有效治疗方法55~57。接受放疗时患者的肠道微生物组的组成,可能是这种并发症的原因。
具体而言,放疗前肠道微生物组多样性低,与放疗后腹泻风险较高有关58。但要了解治疗时微生物组的组成如何影响治疗后的并发症,需要更系统的分析。
为预防或治疗感染,要给接受骨髓移植的癌症患者使用大剂量的抗生素,这会使得他们的肠道微生物组发生剧烈变化。
但是,抗生素是必不可少的,因为在骨髓移植成功前,患者缺乏中性粒细胞,此时发生任何感染都可能会危及生命。
可是,抗生素会对肠道共生微生物造成附带损害:微生物组多样性降低,并且耐万古霉素的细菌(如屎肠球菌)可能会成为优势菌59,从而增加血流感染和患者死亡的风险60。
最近一项关于移植患者的试验表明,自体粪菌移植可以在接受骨髓移植后重建患者的微生物群组(图 6)61。该研究得出下述结论:
自体粪菌移植一般可以恢复患者微生物组的组成,但成功率因人而异。在最佳的情况下,患者几乎恢复了 100%的原始微生物群组成,但在最差的情况下,患者仅恢复了 50%。
成功率差异如此之大的原因尚不清楚,但可能的因素有:用于移植的微生物组的实际组成;患者自体粪菌移植前的微生物组状态;甚至是患者遗传或潜在疾病在内的个人因素。
搞明白为什么粪菌移植的成功率差异巨大,将是未来研究的重要方向。
(之前肠道产业报道过一家进行自体粪菌移植的初创企业:创业 6 年,联手大咖,全球唯一自体菌群移植公司杀入临床 III 期!)
益生菌疗法的效果不仅因为宿主的常驻微生物组而明显不同62,也取决于宿主表达的基因的差异,这表明了微生物组与宿主因素之间复杂的相互作用。
常驻微生物会对益生菌长双歧杆菌的稳定定植产生决定性作用,而这种作用的机制尚不明确63。此外,益生菌会一定程度降低自体粪菌移植的成功率,考虑到这一点是很重要的64。
尽管自体粪菌移植最初成功恢复了造血细胞移植的癌症患者的微生物群,但自体粪菌移植可能对其他癌症效果不佳。如果微生物组在癌症的发生和发展中起关键作用,那么这些患者中接受自体粪菌移植可能会增加复发率。
鉴于我们目前的知识状况,如果不进一步了解微生物组在特定疾病中的作用,自体粪菌移植就不能无风险地用于每种癌症。
我们应该尝试更好地了解肠道中的微生物-微生物、微生物-宿主如何相互作用,来提高益生菌疗法和粪菌移植的成功率,特别是推动基于微生物混合物的个性化癌症疗法65。

图 6 许多癌症疗法,如化疗和盆腔放疗,都会损伤健康的微生物组:
肠道微生物群的移植,例如自体粪菌移植,可用于安全地修复患者的微生物组。自体粪菌移植:收集粪便样品后,清除杂质及耐药菌以确保其安全性,储存到患者微生物群损伤后,移植给患者以提高微生物组多样性。
自体粪菌移植已应用于接受造血细胞移植的癌症患者,但对其他癌症患者的应用应该谨慎进行,因为我们对肠道微生物组与癌症之间的联系不完全了解,而自体粪菌移植可能会引发了一个问题:恢复治疗前微生物组的组成可能会增加癌症复发率。
结束语
人体内和人体表面的众多微生物影响着人体生理的许多方面。因此,似乎合乎逻辑的是,人类微生物组可以影响癌症的预防、发展、治疗和管理,尽管具体机制仍不清楚。
癌症和癌症疗法引起的生理稳态失衡可能会损害微生物组,从而导致更多的并发症。癌症、癌症疗法、肿瘤相关微生物和人类微生物组都可以通过复杂的反馈网络联系起来。破解这些网络及其可塑性需要系统生物学方法。
这里讨论的例子仍未被详尽地研究,但是可以作为科学前沿问题来邀请科学家继续研究,比如可以进一步探究以下问题:
无疑,这个领域受益于微生物组研究的最新进展,从对人类微生物组进行分类学的横向研究,演变为理解功能的纵向研究。微生物组领域的发展使我们能更近一步地理解微生物组在癌症发生和进展中的动态作用。
在癌症-宿主-微生物组这个大型系统中,人类微生物组是其中不可或缺的组成部分。尽管与疾病的严重程度相比,每种单一组成部分似乎都不显著,但最近的研究已经表明,调控微生物组可以将癌症系统推向所需的状态。
如果没有更好的方法在早期预防、检测和治疗癌症,即使发现新的治疗方法,被诊断新患有癌症的患者数量仍将继续超过可用的治疗方法,这意味着我们中的许多人可能需要在有生之年学会与癌症共存。
即使在无法治愈癌症的情况下,若要延长寿命和提高生活质量,也许需要我们利用人类微生物组,提供可持续的、长期控制疾病的方法,这可能有助于使无法治愈的癌症成为一种慢性但易于控制的疾病。
一旦更好地理解这其中的机制,微生物和癌症系统之间的稳态可能最终有助于更健康的生活和更长的寿命。
参考文献:
(滑动下方文字查看)
1.de Martel, C. et al. (2012) Global burden of cancersattributable to infections in 2008: a review and synthetic analysis. LancetOncol. 13, 607–615
2.Turnbaugh, P.J. et al. (2006) An obesity-associatedgut microbiome with increased capacity for energy harvest. Nature 444,1027–1031
3.Ezenwa, V.O. et al. (2012) Animal behavior and themicrobiome. Science 338, 198–199
4.Jobin, C. (2018) Precision medicine using microbiota.Science 359, 32–34
5.Scott, A.J. et al. (2019) International CancerMicrobiome Con- sortium consensus statement on the role of the human microbiomein carcinogenesis. Gut 68, 1624–1632
6.Arends, J. (2010) Metabolism in cancer patients.Anticancer Res. 30, 1863–1868
7.Fujisaka, S. et al. (2018) Diet, genetics, and the gutmicrobiome drive dynamic changes in plasma metabolites. Cell Rep. 22, 3072–3086
8.Ma, C. et al. (2018) Gut microbiome-mediated bile acidmetabo- lism regulates liver cancer via NKT cells. Science 360, eaan5931
9.Kwa, M. et al. (2016) The intestinal microbiome andestrogen receptor-positive female breast cancer. J. Natl. Cancer Inst.108,djw029
10.Baker, J.M. et al. (2017) Estrogen–gut microbiomeaxis: physiological and clinical implications. Maturitas 103, 45–53
11.Devendran, S. et al. (2017) Identification andcharacterization of a 20beta-HSDH from the anaerobic gut bacteriumButyricicoccus desmolans ATCC 43058. J. Lipid Res. 58, 916–925
12.Devendran, S. et al. (2018) The desA and desB genesfrom Clostridium scindens ATCC 35704 encode steroid-17,20-desmolase. J. LipidRes. 59, 1005–1014
13.Zimmermann, M. et al. (2019) Mapping human microbiomedrug metabolism by gut bacteria and their genes. Nature 570, 462–467
14.Schulfer, A.F. et al. (2019) The impact of early-lifesub-therapeutic antibiotic treatment (STAT) on excessive weight is robustdespite transfer of intestinal microbes. ISME J. 13, 1280–1292
15.Wang, Z. et al. (2011) Gut flora metabolism ofphosphatidyl- choline promotes cardiovascular disease. Nature 472, 57–63
16.Carmona-Fontaine, C. et al. (2013) Emergence ofspatial struc- ture in the tumor microenvironment due to the Warburg effect. Proc.Natl. Acad. Sci. 110, 19402–19407
17.Carmona-Fontaine, C. et al. (2017) Metabolic originsof spatial organization in the tumor microenvironment. Proc. Natl. Acad. Sci. U.S. A. 114, 2934–2939
18.Colegio, O.R. et al. (2014) Functional polarizationof tumour-associated macrophages by tumour-derived lactic acid. Nature 513,559–563
19.Sharon, G. et al. (2016) The central nervous systemand the gut microbiome. Cell 167, 915–932
20.Ben-Shaanan, T.L. et al. (2018) Modulation ofanti-tumor immunity by the brain's reward system. Nat. Commun. 9, 2723
21.Alcock, J. et al. (2014) Is eating behaviormanipulated by the gastrointestinal microbiota? Evolutionary pressures andpotential mechanisms. Bioessays 36, 940–949
22.Kiraly, D.D. et al. (2016) Alterations of the hostmicrobiome affect behavioral responses to cocaine. Sci. Rep. 6, 35455
23.Rousseaux, C. et al. (2007) Lactobacillus acidophilusmodu- lates intestinal pain and induces opioid and cannabinoid receptors. Nat.Med. 13, 35–37
24.Sivan, A. et al. (2015) Commensal Bifidobacteriumpromotes antitumor immunity and facilitates anti-PD-L1 efficacy. Science 350,1084–1089
25.Zhou, Y.J. et al. (2017) Cancer killers in the humangut microbiota: diverse phylogeny and broad spectra. Oncotarget 8, 49574–49591
26.Geller, L.T. et al. (2017) Potential role ofintratumor bacteria in mediating tumor resistance to the chemotherapeutic drug gemcitabine.Science 357, 1156–1160
27.Pushalkar, S. et al. (2018) The pancreatic cancermicrobiome promotes oncogenesis by induction of innate and adaptive immunesuppression. Cancer Discov. 8, 403–416
28.Thomas, R.M. et al. (2018) Intestinal microbiotaenhances pancreatic carcinogenesis in preclinical models. Carcinogenesis 39,1068–1078
29.Kostic, A.D. et al. (2012) Genomic analysis identifiesassociation of Fusobacterium with colorectal carcinoma. Genome Res. 22, 292–298
30.Mojica, C.M. et al. (2018) Interventions promotingcolorectal cancer acreening among Latino men: a systematic review. Prev.Chronic Dis. 15, E31
31.Nistal, E. et al. (2015) Factors determiningcolorectal cancer: the role of the intestinal microbiota. Front. Oncol. 5, 220
32.Arthur, J.C. et al. (2012) Intestinal inflammationtargets cancer-inducing activity of the microbiota. Science 338, 120–123
33.Markowitz, S.D. and Bertagnolli, M.M. (2009)Molecular origins of cancer: molecular basis of colorectal cancer. N. Engl. J. Med.361, 2449–2460
34.Coker, O.O. et al. (2019) Enteric fungal microbiotadysbiosis and ecological alterations in colorectal cancer. Gut 68, 654–662
35.Nakatsu, G. et al. (2015) Gut mucosal microbiomeacross stages of colorectal carcinogenesis. Nat. Commun. 6, 8727
36. Nakatsu,G. et al. (2018) Alterations in enteric virome are associated with colorectalcancer and survival outcomes.
Gastroenterology 155, 529–541
37.Yachida, S. et al. (2019) Metagenomic and metabolomicanalyses reveal distinct stage-specific phenotypes of the gut microbiota incolorectal cancer. Nat. Med. 25, 968–976
38.Goncalves, M.D. et al. (2019) High-fructose cornsyrup enhances intestinal tumor growth in mice. Science 363, 1345–1349
39.Ridlon, J.M. et al. (2016) Taurocholic acidmetabolism by gut microbes and colon cancer. Gut Microbes 7, 201–215
40.Dejea, C.M. et al. (2014) Microbiota organization isa distinct feature of proximal colorectal cancers. Proc. Natl. Acad. Sci.
U. S. A. 111, 18321–18326
41.Tomkovich, S. et al. (2019) Human colon mucosalbiofilms from healthy or colon cancer hosts are carcinogenic. J. Clin. Invest.130, 1699–1712
42.Bullman, S. et al. (2017) Analysis of Fusobacteriumpersistence and antibiotic response in colorectal cancer. Science 358, 1443–1448
43.Song, S. et al. (2018) The role of bacteria in cancertherapy -enemies in the past, but allies at present. Infect. Agents Cancer 13,9-9
44.Larkin, J. et al. (2015) Combined nivolumab andipilimumab or monotherapy in untreated melanoma. N. Engl. J. Med. 373, 23–34
45.Gopalakrishnan, V. et al. (2018) Gut microbiome modulates re- sponse toanti-PD-1 immunotherapy in melanoma patients.
Science 359, 97–103
46.Matson, V. et al. (2018) The commensal microbiome isassociated with anti-PD-1 efficacy in metastatic melanoma patients.Science 359,104–108
47.Routy, B. et al. (2018) Gut microbiome influencesefficacy of PD-1-based immunotherapy against epithelial tumors. Science 359,91–97
48.Gharaibeh, R.Z. and Jobin, C. (2019) Microbiota andcancer immunotherapy: in search of microbial signals. Gut 68, 385-288
49.Spor,A. et al. (2011) Unravelling the effects of the environment and host genotypeon the gut microbiome. Nat. Rev. Microbiol. 9, 279–290
50.Frankel, A.E. et al. (2017) Metagenomic shotgun sequencingand unbiased metabolomic profiling identify specific human gut mi- crobiota andmetabolites associated with immune checkpoint therapy efficacy in melanomapatients. Neoplasia 19, 848–855
51.Bachem, A. et al. (2019) Microbiota-derivedshort-chain fatty acids promote the memory potential of antigen-activated CD8+T cells. Immunity 20, 285–297
52.Weber, J.S. et al. (2013) Patterns of onset andresolution of immune-related adverse events of special interest withipilimumab: detailed safety analysis from a phase 3 trial in pa- tients withadvanced melanoma. Cancer 119, 1675–1682
53.Dubin, K. et al. (2016) Intestinal microbiomeanalyses identify melanoma patients at risk for checkpoint-blockade-inducedcolitis. Nat. Commun. 7, 1039112 Trends in Cancer, Month 2020, Vol. xx, No. xx Trendsin Cancer
54.Wang, Y. et al. (2018) Fecal microbiotatransplantation for re- fractory immune checkpoint inhibitor-associatedcolitis. Nat. Med. 24, 1804–1808
55.Benson 3rd, A.B. et al. (2004) Recommended guidelinesfor the treatment of cancer treatment-induced diarrhea. J. Clin. Oncol. 22,2918-2026
56.Morris, K.A. and Haboubi, N.Y. (2015) Pelvicradiation therapy: be- tween delight and disaster. World J. Gastrointest. Surg.7, 279–288
57.Theis, V.S. et al. (2010) Chronic radiationenteritis. Clin. Oncol. (R. Coll. Radiol.) 22, 70–83
58.Wang, A. et al. (2015) Gut microbial dysbiosis maypredict diarrhea and fatigue in patients undergoing pelvic cancer radiotherapy:a pilot study. PLoS One 10, e0126312
59.Taur, Y. et al. (2012) Intestinal domination and therisk of bac- teremia in patients undergoing allogeneic hematopoietic stem celltransplantation. Clin. Infect. Dis. 55, 905–914
60.Taur, Y. et al. (2014) The effects of intestinaltract bacterial diversity on mortality following allogeneic hematopoietic stem celltransplantation. Blood 124, 1174–1182
61.Taur, Y. et al. (2018) Reconstitution of the gutmicrobiota of antibiotic-treated patients by autologous fecal microbiota transplant.Sci. Transl. Med. 10, eaap9489
62.Zmora, N. et al. (2018) Personalized gut mucosalcolonization resistance to empiric probiotics is associated with unique hostand microbiome features. Cell 174, 1388–1405
63.Maldonado-Gomez, M.X. et al. (2016) Stableengraftment of Bifidobacterium longum AH1206 in the human gut depends onindividualized features of the resident microbiome. Cell Host Microbe 20,515–526
64.Suez, J. et al. (2018) Post-antibiotic gut mucosalmicrobiome reconstitution is impaired by probiotics and improved by autologousFMT. Cell 174, 1406–1423
65.Baxter, N.T. et al. (2019) Dynamics of human gutmicrobiota and short-chain fatty acids in response to dietary interventions withthree fermentable fibers. MBio 10, e02566-18
66.Rous, P. (1911) A sarcoma of the fowl transmissibleby an agent separable from the tumor cells. J. Exp. Med. 13, 397–411
67.Stehelin, D. et al. (1976) DNA related to thetransforming gene (s) of avian sarcoma viruses is present in normal avian DNA. Nature260, 170–173
68.Lollini, P.L. et al. (2006) Vaccines for tumourprevention. Nat. Rev. Cancer 6, 204–216
69.Godoy-Vitorino, F. et al. (2018) Cervicovaginal fungiand bacteria associated with cervical intraepithelial neoplasia and high- riskhuman papillomavirus infections in a Hispanic population. Front. Microbiol. 9,2533
70.Rampelli, S. et al. (2016) ViromeScan: a new tool formetagenomic viral community profiling. BMC Genomics 17, 165
71.Garretto, A. et al. (2019) virMine: automateddetection of viral sequences from complex metagenomic samples. PeerJ 7, e6695
72.Barrientos-Somarribas, M. et al. (2018) Discoveringviral ge- nomes in human metagenomic data by predicting unknown proteinfamilies. Sci. Rep. 8, 28
73.Hannigan, G.D. et al. (2018) Diagnostic potential andinteractive dynamics of the colorectal cancer virome. MBio 9, e02248-18
74.Ott, S.J. et al. (2017) Efficacy of sterile fecalfiltrate transfer for treating patients with Clostridium difficile infection.Gastroenterology 152, 799–811
75.Zuo, T. et al. (2018) Bacteriophage transfer duringfaecal microbiota transplantation in Clostridium difficile infection is associatedwith treatment outcome. Gut 67, 634–643
76.Hourigan, S.K. et al. (2019) Fecal transplant inchildren with Clostridioides difficile gives sustained reduction inantimicrobial resistance and potential pathogen burden. Open Forum Infect. Dis.6, ofz379
77.Amieva, M. and Peek Jr., R.M. (2016) Pathobiology of Helicobacterpylori-Induced Gastric Cancer. Gastroenterology 150, 64–78
78.Amiri, M. et al. (2011) The decline in stomach cancermortality: exploration of future trends in seven European countries. Eur. J.Epidemiol. 26, 23–28
79.Linz, B. et al. (2007) An African origin for theintimate association between humans and Helicobacter pylori. Nature 445,915–918
80.Blaser, M.J. and Atherton, J.C. (2004) Helicobacter pyloripersistence: biology and disease. J. Clin. Invest. 113, 321–333 81. Chen, C. etal. (2017) Accumulated evidence on Helicobacter pylori infection and the risk ofasthma: a meta-analysis. Ann. Allergy Asthma Immunol. 119, 137–145
82.Kira, J.I. and Isobe, N. (2019) Helicobacter pyloriinfection and demyelinating disease of the central nervous system. J.Neuroimmunol. 329, 14–19
83.Piovani, D. et al. (2019) Environmental risk factorsfor inflammatory bowel diseases: an umbrella review of meta-analyses. Gastroenterology157, 647–659
84.Gravina, A.G. et al. (2018) Helicobacter pylori andextragastric diseases: a review. World J. Gastroenterol. 24, 3204–3221 85.Hosseininasab Nodoushan, S.A. and Nabavi, A. (2019) The interaction ofHelicobacter pylori infection and type 2 diabetes mellitus. Adv. Biomed. Res.8, 15
86.Dzutsev, A. et al. (2017) Microbes and cancer. Annu.Rev. Immunol. 35, 199–228
87.Redelman-Sidi, G. et al. (2014) The mechanism ofaction of BCG therapy for bladder cancer – a current perspective. Nat.Rev.Urol. 11, 153–162
88.Earle, K.A. et al. (2015) Quantitative Imaging of gutmicrobiota spatial organization. Cell Host Microbe 18, 478–488
89.Tropini, C. et al. (2017) The gut microbiome:connecting spatial organization to function. Cell Host Microbe 21, 433–442
90.Mark Welch, J.L. et al. (2017) Spatial organizationof a model 15-member human gut microbiota established in gnotobiotic mice.Proc. Natl. Acad. Sci. U. S. A. 114, E9105–E9114
91.Moffitt, J.R. et al. (2016) High-performancemultiplexed fluores- cence in situ hybridization in culture and tissue withmatrix imprinting and clearing. Proc. Natl. Acad. Sci. U. S. A. 113, 14456–14461
92.Ghaffarizadeh, A. et al. (2018) PhysiCell: an opensource physics-based cell simulator for 3-D multicellular systems. PLoS Comput.Biol. 14, e1005991
93.Ward, T. et al. (2017) BugBase predictsorganism-level microbiome phenotypes. bioRxiv. Published online May 7, 2017.https://doi.org/10.1101/133462
94.Douglas, G.M. et al. (2019) PICRUSt2: an improved andextensible approach for metagenome inference. bioRxiv. Published online June15, 2019. https://doi.org/10.1101/672295
95.Bolyen, E. et al. (2019) Reproducible, interactive,scalable and extensible microbiome data science using QIIME 2. Nat. Biotechnol.37, 852–857
96.Xavier, J.B. et al. (2007) Multi-scaleindividual-based model of microbial and bioconversion dynamics in aerobicgranular sludge. Environ. Sci. Technol. 41, 6410–6417
97.Pasolli, E. et al. (2019) Extensive unexplored humanmicrobiome diversity revealed by over 150,000 genomes from metagenomes spanningage, geography, and lifestyle. Cell 176, 649–662.e20
98.Smits, S.A. et al. (2017) Seasonal cycling in the gutmicrobiome of the Hadza hunter-gatherers of Tanzania. Science 357, 802–806
99.Belkaid, Y. and Hand, T.W. (2014) Role of themicrobiota in immunity and inflammation. Cell 157, 121–141
100.David, L.A. et al. (2014) Diet rapidly andreproducibly alters the human gut microbiome. Nature 505, 559–563
101.Chassaing, B. et al. (2015) Dietary emulsifiersimpact the mouse gut microbiota promoting colitis and metabolic syndrome.Nature 519, 92–96
102.Viennois, E. et al. (2017) Dietaryemulsifier-induced low-grade inflammation promotes colon carcinogenesis. CancerRes.77, 27–40
103.Scudellari, M. (2017) Cleaning up the hygienehypothesis. Proc.Natl. Acad. Sci. U. S. A. 114, 1433–1436
104.Vangay, P. et al. (2018) US immigration westernizesthe human gut microbiome. Cell 175, 962–972
105.Bello, M.G.D. et al. (2018) Preserving microbialdiversity. Science 362, 33–34
106.Rohlke, F. and Stollman, N. (2012) Fecal microbiotatransplantation in relapsing Clostridium difficile infection. Ther. Adv.Gastroenterol. 5, 403–420
原文链接:https://doi.org/10.1016/j.trecan.2020.01.004
作者|Joao B. Xavier 等人
编译|赵婧
审校|617


