
第四章 肠道微生物组研究14个主题方向
4.4 动物
4.4.2 主要认知和研究现状
4.4.2.2 养殖动物
反刍动物
反刍动物胃肠道微生物包括细菌、真核生物、古菌和病毒等,以细菌为主,其中厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)和纤维杆菌门(Fibrobacteres)约占67%,柔膜菌门(Tenericutes)和放线菌门(Actinobacteria)等也占一定比例[29],目前重建了10,373个非冗余的微生物基因组且在种水平上鉴定出8745个未培养的候选细菌和古菌物种[30]。瘤胃菌群是反刍动物消化系统的组成部分,核心细菌门为厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),包括纤维素降解菌(如产琥珀酸丝状杆菌)、半纤维素降解菌(如普雷沃氏菌)等[31]。

图4.4.11 反刍动物胃肠道微生物组组成的多样性(上图)和功能(下图)[30]
古菌是反刍动物肠道微生物群的常见组成成分,多样性、丰度受宿主、饮食类型、纤维含量和肠道生理等因素影响[32],最丰富的类群是甲烷杆菌目(Methanobacteriales),见图1.1.10。瘤胃古菌主要由产甲烷微生物组成,被称为产甲烷菌,主要包括甲烷短杆菌属(Methanobrevibacter)、甲烷球菌属(Methanosphaera)、甲烷八叠球菌属(Methanosarcina)、广古菌目(Methanomassiliicoccales)[33]。这些古菌参与食物消化过程并产生甲烷等代谢产物,内共生的产甲烷古菌可利用由丙酮酸氧化产生的氢气生成甲烷,而甲烷主要通过反刍动物的打嗝和排气排放。反刍动物的胃肠道含有丰富的病毒组,其中噬菌体是肠道病毒组的主要成员,约60%的噬菌体为裂解型[34]。其中瘤胃病毒组可调节瘤胃微生物组,大多数可分类的vOTUs注释为有尾噬菌体目(Caudovirales)[35]。
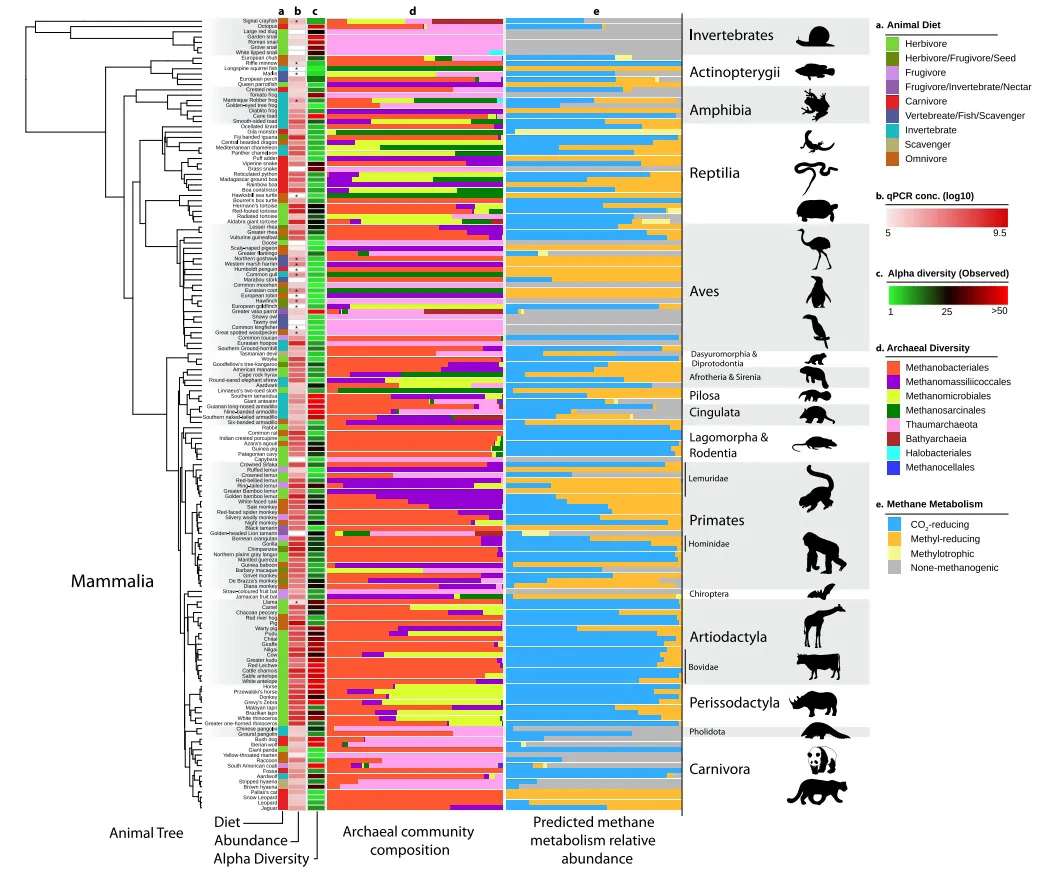
图4.4.12 反刍动物肠道中的古菌组成丰度和多样性[32]
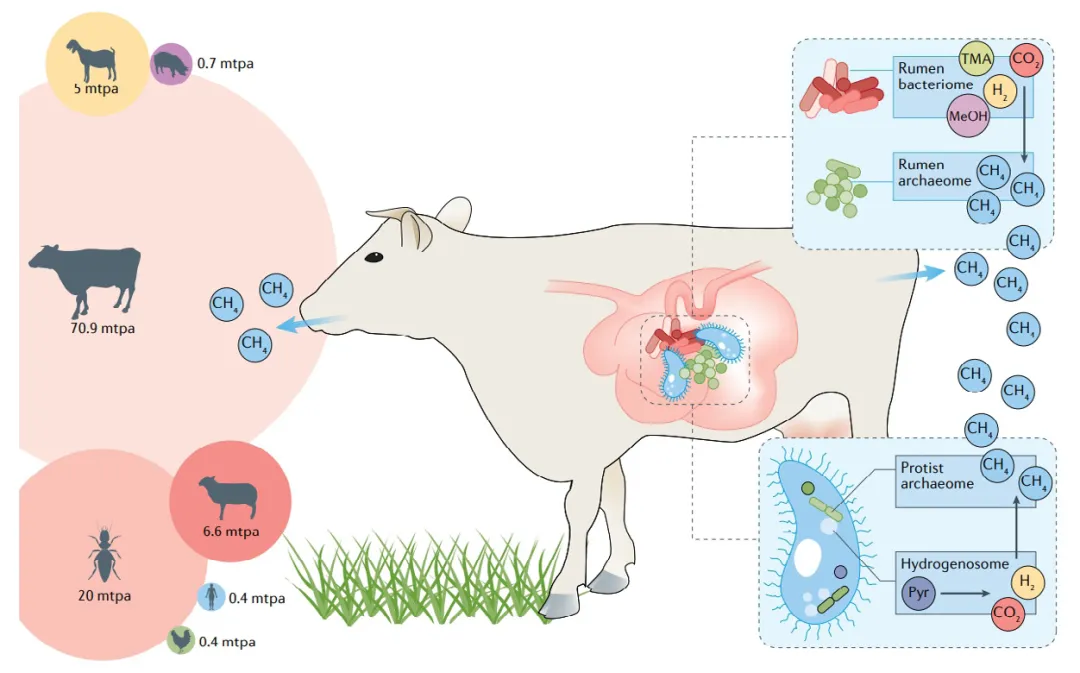
图4.4.13 牛与其他动物的甲烷排放对比[33]
反刍动物能广泛利用高纤维饲料,主要依靠瘤胃细菌降解。瘤胃微生物可以帮助宿主利用纤维类物质以及发酵产生挥发性脂肪酸和氢化脂肪酸等,其中挥发性脂肪酸是宿主的主要能量来源。此外,维生素B族、维生素K2合成主要由瘤胃菌群完成。虽然大肠菌群也有一定比例的贡献,但更多参与的是多糖和氨基酸代谢[36]。
从功能角度上看,反刍动物的胃肠道微生物组主要可以划分为3个组,分别为胃、小肠和大肠(图4.4.11)。碳水化合物活性酶(CAZyme)广泛存在于不同类型的消化道细菌,其中厚壁菌门、拟杆菌门是最主要的生产者。瘤胃微生物主要编码纤维素酶、半纤维素酶和果胶酶,小肠微生物主要编码溶菌酶和几丁质酶。反刍动物的胃肠道微生物组大约含有60%微生物基因组编码氢酶基因,具有代谢氢的能力。另外,50%微生物基因组能通过发酵途径产生氢分子[30]。
猪
猪的肠道菌群包括细菌、真菌和病毒等微生物,其中细菌占98.9%以上,其余属于病毒和古菌。细菌主要来自厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes),占肠道细菌总数的70%以上,其次是变形菌门(Proteobacteria)和放线菌门(Actinobacteria)[37]。猪肠道的优势菌属包括拟杆菌属(Bacteroides)、埃希氏菌属(Escherichia)、普雷沃氏菌属(Prevotella)、链球菌属(Streptococcu)、乳酸菌属(Lactobacillus)等[38]。猪肠道的核心菌种主要由罗伊氏乳杆菌(Lactobacillus reuteri)、约氏乳杆菌(Lactobacillusjohnsonii)、淀粉乳杆菌(Lactobacillus amylovorus)、Ruminococcaceae bacterium、大肠杆菌(Escherichiacoli)、Prevotella copri、脆弱拟杆菌(Bacteroides fragilis)、猪链球菌(Streptococcus suis)、Phascolarctobacterium succinatutens、肠沙门氏菌(Salmonella enterica)等组成[37]。
猪肠道微生物功能主要包括:氨基酸生物合成和代谢,次生胆汁酸生物合成、脂肪酸生物合成和降解等脂质代谢,碳水化合物代谢,维生素B6、生物素代谢等维生素代谢,新霉素、卡那霉素和庆大霉素生物合成等抗生素生物合成,DNA复制、同源重组等遗传相关途径信息处理。此外,降解淀粉、果聚糖和乳糖的细菌可以帮助仔猪从母乳到固体饲料的转变,增强了从膳食碳水化合物获取能量的能力[39]。
家禽
家禽消化道由嗉囊、腺胃、肌胃、小肠、盲肠、结肠和泄殖腔等组成,定植着细菌、病毒、真菌等微生物。相比哺乳动物,家禽消化道较短,食物停留时间较短,因此肠道微生物的数量和多样性较低。家禽的肠道细菌主要以厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)为主[40],占菌群总量的90%以上。家禽优势菌属主要包括Phocaeicola、拟杆菌属(Bacteroides)、另枝菌属(Alistipes)、普雷沃氏菌属(Prevotella)、埃希氏菌属(Escherichia)等(图4.4.14)。鸡肠道真菌主要分布于4个门,以子囊菌门(Ascomycota)、担子菌门(Basidiomycota) 为主, 占真菌菌群的90% 以上,属水平则以赤霉属(Gibberella)、曲霉属(Aspergillus)、念珠菌属(Candida)最丰富[41]。

图4.4.14 江汉鸡肠道菌群分类[42]
家禽肠道可分为前肠和后肠,前肠包含十二指肠、空肠和回肠,后肠包含盲肠和结直肠。前肠微生物的功能主要包括遗传信息处理(复制和修复、翻译和转录)、及核苷酸和脂质代谢,后肠微生物的功能则以氨基酸代谢、能量代谢和次级代谢产物的生物合成为主[40]。
水生生物
家禽消化道由嗉囊、腺胃、肌胃、小肠、盲肠、结肠和泄殖腔等组成,定植着细菌、病毒、真菌等微生物。相比哺乳动物,家禽消化道较短,食物停留时间较短,因此肠道微生物的数量和多样性较低。家禽的肠道细菌主要以厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、变形菌门(Proteobacteria)为主[40],占菌群总量的90%以上。家禽优势菌属主要包括Phocaeicola、拟杆菌属(Bacteroides)、另枝菌属(Alistipes)、水生动物的肠道菌群主要包括需氧微生物、兼性厌氧微生物和专性厌氧微生物,优势菌门为变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、蓝细菌门(Cyanobacteria), 见图4.4.15,宿主栖息地和身体部位是肠道微生物群的主要决定因素[43,44]。水生生物的肠道主要以弧菌科(Vibrionaceae)的发光杆菌属(Photobacterium)为主。在双斑红鲷鱼,肠道弧菌科(Vibrionaceae)细菌的数量远超其他科细菌[45]。

图4.4.15 鱼类样本肠道微生物群细菌门相对丰度[44]
水生动物肠道菌群的最主要基因功能包括碳水化合物相关,氨基酸及衍生物相关,蛋白质代谢,辅酶因子、维生素、修复组和色素相关,以及DNA代谢等,共生细菌的蛋白代谢功能强大,细胞质富含核糖体蛋白[45]。
聚焦:靶向动物微生物组干预
对动物肠道微生物组研究的不断深入,揭示了消化道微生物在养殖动物领域所发挥的重要作用。近年来,多项研究通过靶向微生物组干预养殖动物的生长发育和健康水平[46-51]。益生菌、益生元、粪菌移植(FMT)改善了养殖动物的腹泻、乳腺炎和炎症等问题,显著提高了生产性能。这些成果表明调控肠道菌群结构干预养殖动物具有非常广阔的应用前景。
近期代表性研究

聚焦:助力动物养殖
对动物肠道微生物组研究的不断深入,揭示了消化道微生物在养殖动物领域所发挥的重要作用。近年来,多项研究通过靶向微生物组干预养殖动物的生长发育和健康水平[46-51]。益生菌、益生元、粪菌移植(FMT)改善了养殖动物的腹泻、乳腺炎和炎症等问题,显著提高了生产性能。这些成果表明调控肠道菌群结构干预养殖动物具有非常广阔的应用前景。饲料添加抗生素会严重破坏动物肠道的微生态平衡,且易在动物体内残留,严重影响动物产品品质和人类健康。随着对动物产品安全性问题的日益关注,人们将寻找抗生素的替代物来调节肠道微生态视为一项重要的策略,因为其可以改善动物的生长性能并对抗动物疾病,同时减少抗生素的使用对环境和公共健康的影响。近期有关肠道菌群对动物养殖、生长发育的影响以及生物学机制的研究取得了诸多进展[52-55],其中更多集中于益生菌改善动物的健康和品质。
近期代表性研究

4.4.2.3 野生动物
大熊猫
大熊猫是全球易危物种,肠道细微生物以厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)为主,其次是拟杆菌门(Bacteroidota)、放线菌门(Actinobacteriota)。大熊猫肠道最丰富的5个属分别是链球菌属(Streptococcus)、梭菌属(Clostridium)、埃希氏菌属(Escherichia)、明串珠菌属(Leuconostoc)、假单胞菌E属(Pseudomonas E)。最丰富的MAG为非解乳糖链球菌(Streptococcus alactolyticus)。(图4.4.16)

图4.4.16 大熊猫微生物基因组的最大似然树[58]
大熊猫的肠道病毒组含有丰富的噬菌体,且不同年龄阶段的大熊猫幼仔及成年大熊猫不同。成年大熊猫的粪便样本中针对柠檬酸杆菌属、阪崎肠杆菌、果胶杆菌属、欧文氏菌属、Dickeya、肠球菌属及假单胞菌属的噬菌体丰度更高,大熊猫幼仔中针对乳球菌属、链球菌属、乳杆菌属、明串珠菌属的噬菌体丰度更高。主要噬菌体物种的丰度及多样性随年龄增长而升高[56]。
大熊猫的饮食完全由高度纤维的竹子组成,却呈现典型的食肉动物样胃肠道特征。大熊猫的肠道微生物功能主要包括碳水化合物代谢、氨基酸代谢、脂肪酸代谢、能量代谢、膜转运和遗传信息处理等[57]。虽然大熊猫的基因组缺乏消化纤维素和半纤维素所需的酶,但是肠道存在能够消化纤维素、半纤维素等营养物质的细菌,并含有半纤维素消化酶和纤维素酶相关编码基因。与食草动物和杂食动物相比,大熊猫的肠道微生物组参与碳水化合物代谢的基因较低,但参与蛋白质代谢的基因在丰度和表达上更高。从大熊猫分离出的非解乳糖链球菌灌胃小鼠后,可显著增加小鼠空肠的必需氨基酸相对丰度[58]。
灵长类
灵长类动物的肠道含有数以万亿计的细菌,有助于新陈代谢、免疫系统发育和抵抗病原体。灵长类动物的肠道微生物主要以厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidota)为主,其次是螺旋体门(Spirochaetes)、变形菌门(Proteobacteria)[59]。灵长类动物肠道的优势菌科为毛螺菌科(Lachnospiraceae)、螺旋体科(Spirochaetaceae)、普雷沃氏菌科(Prevotellaceae)、颤螺旋菌科(Oscillospiraceae)、瘤胃球菌科(Ruminococcaceae),密螺旋体属(Treponema)、普雷沃氏菌属(Prevotella)是两个最丰富的类群。此外,肠道相关原生生物及线虫广泛分布于非人灵长类物种,与细菌不同,真核生物的组成表现为宿主物种特异性较低,且很少受到饮食的影响[60]。
灵长类动物的肠道菌群有助于宿主适应不同的饮食和环境变化,会随季节性降雨模式和主食果实类型波动。灵长类动物的肠道微生物的核心功能,主要包括碳水化合物代谢、氨基酸代谢、辅助因子和维生素代谢、能量代谢、膜转运和遗传信息处理等。在雨季,参与细菌膜转运、DNA复制、DNA修复以及细胞运动途径增加,还有淀粉和蔗糖代谢,果糖,甘露糖和半乳糖等糖代谢增加。在干燥时期,参与能量、氨基酸和脂质代谢的细菌基因增加,能量代谢途径增加包括柠檬酸循环、氧化磷酸化以及脂肪酸合成和代谢途径,还有甲烷途径和碳固定途径,与蛋白质氨基酸合成、蛋白质的翻译、合成以及脂多糖合成的相关功能也增加[61]。
聚焦:野化放归路
野生动物在人工圈养种群增长到一定数量之后,需要将部分人工圈养动物放归野外,进而改善其遗传多样性、消除种群灭绝风险。近年一些研究探索了野生动物肠道微生物组成和功能的多样性,比较了圈养种群和野生种群的肠道菌群,为野化放归路提供了一定的基础[62-64]。
近期代表性研究

聚焦:环境压力与疾病传播
细菌传播是双向的、跨物种的,栖息地人为干扰、生境破碎化、宿主生态和人类活动均会导致细菌传播,而有针对性地干预可以有效阻止疾病传播、降低紧急事件的发生率。这种方法不仅为控制病菌传播提供了新的途径,也有助于人类健康和野生动物保护。野生动物肠道菌群的动态变化可作为评估栖息地质量、食物利用状况及野生动物健康的指标,有助于未来野生动物保护[65-68]。
近期代表性研究

4.4.2.4 宠物
聚焦:改善宠物健康
宠物是人们日常生活的重要伙伴,常被视为家庭的一部分。年龄、品种、饮食组成、疾病、环境、食物类型和性别等因素都会影响宠物的微生物群组成。宠物肠道微生物主要以拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、梭杆菌门(Fusobacteria)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)为主。厚壁菌门(Firmicutes)在猫胃肠道菌群占主导地位,,变形菌门(Proteobacteria)在犬肠道更丰富[69]。在目水平,猫肠道菌群以梭菌目(Clostridiales)占主导地位,其次是乳杆菌目(Lactobacillales)、拟杆菌目(Bacteroidales)、弯曲菌目(Campylobacterales)、梭杆菌目(Fusobacteriales),见图4.4.17。在属水平,犬肠道以乳酸菌属(Lactobacillus)占主导地位,其他是双歧杆菌属(Bifidobacterium)、肠球菌属(Enterococcus)、链球菌属(Streptococcus)、片球菌属(Pediococcus)等,见图4.4.18。

图4.4.17 猫鼻腔、口腔和肠道菌群分布[69]

图4.4.18 犬鼻腔、口腔和肠道菌群分布[69]
宠物肠道微生物的核心功能主要包括碳水化合物代谢、辅助因子代谢、维生素代谢及能量代谢等。碳水化合物代谢包括与甘露糖、寡糖和棉子糖代谢相关的代谢,微生物对碳水化合物的发酵产生短链脂肪酸(宿主能量来源)。维生素K和维生素B合成也是肠道微生物群的重要功能。
聚焦:改善宠物健康
近年来宠物行业高速发展,健康问题也凸显出来。研究表明,48.4%的宠物犬、52.8%的宠物猫存在健康问题。宠物的肠道微生物组被认为是与宠物健康密切相关的的代谢器官,近年有研究从肠道微生物组着力于改善宠物健康[70]。
近期代表性研究

参考文献
29.Cholewińska, P., et al., The microbiome of the digestive system of ruminants - a review. Anim Health Res Rev, 2020. 21(1): p.3-14.
30.Xie, F., et al., An integrated gene catalog and over 10,000 metagenome-assembled genomes from the gastrointestinal microbiome of ruminants. Microbiome, 2021. 9(1): p. 137.
31.Mizrahi, I., R.J. Wallace, and S. Moraïs, The rumen microbiome: balancing food security and environmental impacts. Nat Rev Microbiol, 2021. 19(9): p. 553-566.
32.Thomas, C.M., et al., Factors shaping the abundance and diversity of the gut archaeome across the animal kingdom.Nature Communications, 2022. 13(1): p. 3358.
33.Borrel, G., et al., The host-associated archaeome. Nature Reviews Microbiology, 2020. 18(11): p. 622-636.
34.Wu, Y., et al., A compendium of ruminant gastrointestinal phage genomes revealed a higher proportion of lytic phages than in any other environments. Microbiome, 2024. 12(1): p. 69.
35.Yan, M., et al., Interrogating the viral dark matter of the rumen ecosystem with a global virome database. NatureCommunications, 2023. 14(1): p. 5254.
36.Jiang, Q., et al., Metagenomic insights into the microbe-mediated B and K(2) vitamin biosynthesis in the gastrointestinal microbiome of ruminants. Microbiome, 2022. 10(1): p. 109.
37.Chen, C., et al., Expanded catalog of microbial genes and metagenome-assembled genomes from the pig gut microbiome. Nature Communications, 2021. 12(1): p. 1106.
38.He, M., et al., Evaluating the Contribution of Gut Microbiota to the Variation of Porcine Fatness with the Cecum and Fecal Samples. Front Microbiol, 2016. 7: p. 2108.
39.Wang, W., et al., Metagenomic reconstructions of gut microbial metabolism in weanling pigs. Microbiome, 2019. 7(1): p. 48.
40.Huang, P., et al., The chicken gut metagenome and the modulatory effects of plant-derived benzylisoquinoline alkaloids. Microbiome, 2018. 6(1): p. 211.
41.Robinson, K., et al., Biogeography, succession, and origin of the chicken intestinal mycobiome. Microbiome, 2022. 10(1): p.55.
42.Shen, H., et al., Metagenome-assembled genome reveals species and functional composition of Jianghan chicken gut microbiota and isolation of Pediococcus acidilactic with probiotic properties. Microbiome, 2024. 12(1): p. 25.
43.Minich, J.J., et al., Host biology, ecology and the environment influence microbial biomass and diversity in 101 marine fish species. Nat Commun, 2022. 13(1): p. 6978.
44.Kim, P.S., et al., Host habitat is the major determinant of the gut microbiome of fish. Microbiome, 2021. 9(1): p. 166.
45.Le Doujet, T., et al., Closely-related Photobacterium strains comprise the majority of bacteria in the gut of migrating Atlanticcod (Gadus morhua). Microbiome, 2019. 7(1): p. 64.
46.Chen, C., et al., Prevotella copri increases fat accumulation in pigs fed with formula diets. Microbiome, 2021. 9(1): p. 175.
47.Wang, Y., et al., Dietary Supplementation of Inulin Ameliorates Subclinical Mastitis via Regulation of Rumen Microbial Community and Metabolites in Dairy Cows. Microbiol Spectr, 2021. 9(2): p. e0010521.
48.Chen, Y., et al., Chicken cecal microbiota reduces abdominal fat deposition by regulating fat metabolism. npj Biofilms and Microbiomes, 2023. 9(1): p. 28.
49.Zhao, C., et al., Commensal cow Roseburia reduces gut-dysbiosis-induced mastitis through inhibiting bacterialtranslocation by producing butyrate in mice. Cell Rep, 2022. 41(8): p. 111681.
50.Zhang, X., et al., Chicken jejunal microbiota improves growth performance by mitigating intestinal inflammation.Microbiome, 2022. 10(1): p. 107.
51.Kim, H.S., et al., Longitudinal evaluation of fecal microbiota transplantation for ameliorating calf diarrhea and improving growth performance. Nat Commun, 2021. 12(1): p. 161.
52.Yin, J., et al., Obese Ningxiang pig-derived microbiota rewires carnitine metabolism to promote muscle fatty acid deposition in lean DLY pigs. Innovation (Camb), 2023. 4(5): p. 100486.
53.Tang, S., et al., Xylooligosaccharide-mediated gut microbiota enhances gut barrier and modulates gut immunity associated with alterations of biological processes in a pig model. Carbohydr Polym, 2022. 294: p. 119776.
54.Brunse, A., et al., Fecal filtrate transplantation protects against necrotizing enterocolitis. Isme j, 2022. 16(3): p. 686-694.
55.Chen, X., et al., Effects of Dietary Supplementation with Lactobacillus acidophilus and Bacillus subtilis on Mucosal Immunity and Intestinal Barrier Are Associated with Its Modulation of Gut Metabolites and Microbiota in Late-Phase Laying Hens.Probiotics Antimicrob Proteins, 2023. 15(4): p. 912-924.
56.Guo, M., et al., Dynamics of bacteriophages in gut of giant pandas reveal a potential regulation of dietary intake on bacteriophage composition. Sci Total Environ, 2020. 734: p. 139424.
57.Huang, G., et al., PandaGUT provides new insights into bacterial diversity, function, and resistome landscapes with implications for conservation. Microbiome, 2023. 11(1): p. 221.
58.Deng, F., et al., The unique gut microbiome of giant pandas involved in protein metabolism contributes to the host's dietary adaption to bamboo. Microbiome, 2023. 11(1): p. 180.
59.Sadoughi, B., et al., Aging gut microbiota of wild macaques are equally diverse, less stable, but progressively personalized.Microbiome, 2022. 10(1): p. 95.
60.Mann, A.E., et al., Biodiversity of protists and nematodes in the wild nonhuman primate gut. The ISME Journal, 2020. 14(2): p.609-622.
61.Baniel, A., et al., Seasonal shifts in the gut microbiome indicate plastic responses to diet in wild geladas. Microbiome, 2021.9(1): p. 26.
62.Diaz, J., K.H. Redford, and A.T. Reese, Captive and urban environments are associated with distinct gut microbiota in deer mice (Peromyscus maniculatus). Biol Lett, 2023. 19(3): p. 20220547.
63.Zhou, W., et al., Symbiotic bacteria mediate volatile chemical signal synthesis in a large solitary mammal species. Isme j,2021. 15(7): p. 2070-2080.
64.Campbell, T.P., et al., The microbiome and resistome of chimpanzees, gorillas, and humans across host lifestyle and geography. Isme j, 2020. 14(6): p. 1584-1599.
65.Jernfors, T., et al., Association between gut health and gut microbiota in a polluted environment. Sci Total Environ, 2024.914: p. 169804.
66.He, W.T., et al., Virome characterization of game animals in China reveals a spectrum of emerging pathogens. Cell, 2022.185(7): p. 1117-1129.e8.
67.Chen, Y.M., et al., Host traits shape virome composition and virus transmission in wild small mammals. Cell, 2023. 186(21): p.4662-4675.e12.
68.Levin, D., et al., Diversity and functional landscapes in the microbiota of animals in the wild. Science, 2021. 372(6539).
69.Lee, D., et al., Perspectives and advances in probiotics and the gut microbiome in companion animals. J Anim Sci Technol,2022. 64(2): p. 197-217.
70.Ma, X., et al., Whole-Genome Shotgun Metagenomic Sequencing Reveals Distinct Gut Microbiome Signatures of Obese Cats.Microbiol Spectr, 2022. 10(3): p. e0083722.



