
各位朋友,大家晚上好!
我是来自中国科学院北京生命科研院的赵方庆,下面由我给大家分享一下我们实验室做的一些工作,包括菌群的分析。
我相信在座的各位来参加“肠·道”论坛,意味着对菌群感兴趣。
大家可能都非常关注菌群如何影响健康,菌群到底有什么样的作用?
大家可能不知道的是,如何获得菌群的这些数据和信息,如何了解菌群和健康之间的关系?

为了更好地展示这一点,我提出来一个比较形象化的类比,可以把菌群想象成一种江湖。
大家都看过武侠小说,在江湖里面有好多的门派有好多武术招数,同样对于菌群也有分类的阶元,比如说门、纲、目、科、属、种。
江湖有峨眉派、有武当派等等,这些的门派都有自己的一些独门秘籍,如打狗棒法、玉女心经等等。
同样的,菌群也有自己特殊的一些地方,某些物种它编码特定的一些基因,具有一些特定的功能,这些基因和功能,可能影响我们的健康,导致相应的一些疾病。
另外,在江湖当中不同的帮派之间可能有竞争、有联盟,也有一些勾心斗角。同样对于菌群,它们的关系也是非常复杂的,这里面有互利共生,也有竞争拮抗。
如何去研究和揭示这些菌群与我们健康和疾病之间的关系呢?其实这就需要对我们这些菌群有着更为深入的一些了解。
我们经常会提到微生物的暗物质,这是因为我们过多地关注人体自身的基因,而忽略了和我们共生的这些菌群,同样的我们居于庙堂之高,可能也忽略了江湖之远。

下面我就给大家介绍下,如何真正地去了解我们身边以及我们肠道之内的这些微生物,如何来做好一个江湖百晓生,对这些与我们共生的细菌了如指掌。
大家应该都看过《天龙八部》,都知道神仙姐姐王语嫣,她博闻强记,把各个门派的这些武功、招数都记到自己脑海里。
她可以从一个简单的招数里面推断这个人,他是什么帮派以及他下一步、下一个招数可能要使什么招,可以说她就是武侠界的 AlphaGo。
那么做菌群的分析,也需要具有这样的一些本领。我们需要识别某些特定的 Marker 基因,就像是一些招数似的,从它第一步做了什么就可以推断后面是什么表现。
比如可以测定它的核糖体 RNA 的序列,通过这个序列本身的一些特点,可以给它一个具体的分类的阶元,它属于什么种、属,它的丰度是什么?

当然如果关注它的功能的话,必须从这种简单的分类,推向对它功能基因的认知。比如可以通过测定当前菌群全基因组的序列,分析它的功能基因,去推断当前这个物种它的功能以及是如何影响我们健康的。
这里面就存在一个问题,如何来揭示真正和疾病有关的这些微生物、这些株系。

一般情况下,我们可能去测定一个16S,来判断哪些种或者哪些属和健康有关。
但这时候还缺乏具体可操作的手段,仍然需要找到某一个特定的细菌、一个细胞、一个株系,去做后续的功能的一些研究,这里面又牵扯到因果关系转向关联关系的这样的一个转变。
目前非常需要相应的一些方法、一些技术手段来找到感兴趣的特定的微生物、特定的基因和特定的变异。
这就需要一些更为有效的生物大数据的挖掘方法,包括如何去精准地解读海量的、零散的数据信息。除此之外,也需要靶向地去分析的这样一些方法。
比如现在测序得到一大堆海量的序列,它的数据量非常非常大,即使借助最先进的超算的一些技术,仍然可能难以把所有的信息都给挖掘出来,那么就需要做靶向,把你关心的某一个特定的微生物其特定的基因功能通过建立相应的方法给找出来。
我们团队在过去的几年之内,就从微生物的谱系的筛查,到功能基因的重建,然后建立了一系列的方法。
这里面我们非常关心的是和人体健康有关的这些菌群,它们具有什么样的功能、基因,如何来重建它的基因的序列。

也就意味着我们要去解读各个门派或者各个团体,它们具有什么样的独门秘籍以及这个秘籍彼此之间的关联是什么样子。

目前如何去研究相应的这种功能基因,如何从海量的组学的数据微生物组的样本当中,来筛选来获得它的精细的基因组图谱?
大家提了一系列的方法,包括最近大家所关心的采用单细胞的技术来获得每一个细胞的基因组。
2013年,就有科学家用了这样一些方法,通过从不同环境来筛选微生物的单细胞,通过建库、测序最终获得很多微生物的全基因组序列。
这些序列揭示与我们共生的菌群以及揭示一些新的化合物、新的基因、新的功能,提供了非常好的一些条件。
但是这些方法都受限于单细胞技术的低效性以及高度的偏倚性。
具体说,就是由于单细胞测序技术本身只能得到部分的,大概一半左右的基因组的全序列,也就意味着很多的它对应的核酸序列通过扩增之后完全丢失了。

大家就想,对于一个单细胞,它可能只含有飞克级的核酸,如何来增加核酸的拷贝数?
大家想了一系列的方法,包括通过某些特定的一些化合物,来抑制细胞的分裂。
本来一个细胞随着细胞的分裂它里边的核酸会复制,也会平均分配到两个子代细胞当中,如果把细胞的分裂给抑制住了,它只有核酸的加倍并没有细胞的分裂。通过测定这一个没有分裂的细胞的核酸,就可以获得更为完整的基因组序列。
同样还有一些类似的方法,包括采用捕获的方法,把特定的单细胞给局限在某一个特定的一些微球或者孔洞里面,通过局部的增殖就可以获得大量的从单细胞所发展出来的多个克隆,以及相应的这种基因组的信息。
这些方法,都从实验技术上,提高了研究这些基因组的这样一些手段。
除了这些实验的手段之外,生物信息学家也提出来一系列的方法,采用计算的方法、生物大数据挖掘的方法来帮助我们去解析微生物组的数据。
比如今天在座可能有二、三百位听众,大家每个人肠道里面可能有成百上千个不同种类的微生物,如果对某个个体的菌群来进行测定,显然这个数据的解析是非常非常困难。
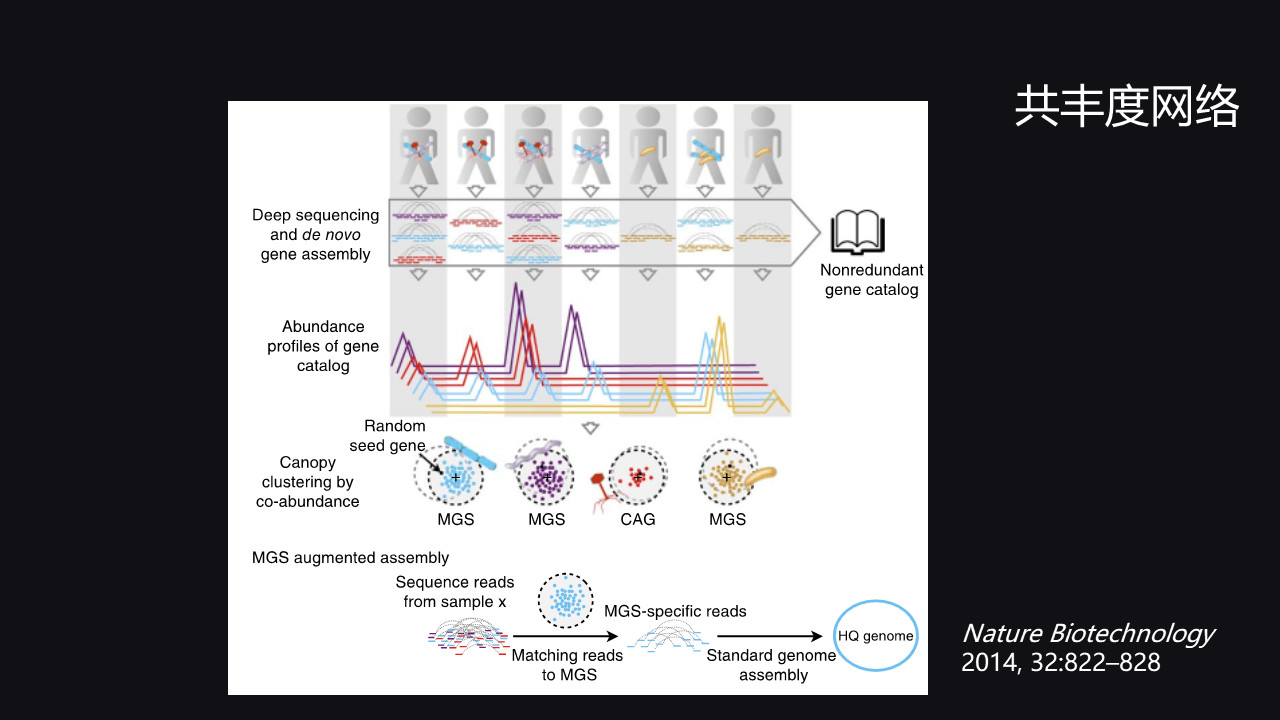
如果把所有人的菌群都进行解析,由于某一个菌在所有的人里面可能都有,但是它的丰度不一样,利用这种丰度的差异,就可以把来源于同一个基因组的这些序列通过聚类的方法聚到一起。
这个方法称为共丰度,是一种网络重建的方法。利用这个方法,就可以基于没有参考基因组的情况下,来获得肠道或者其他环境当中的微生物组的序列。
这个方法很显然就依赖于海量的数据的处理,首先要测定所有人的肠道微生物,所以它的成本和花费都是非常高的。
我们就想,能不能借助于单细胞测序,加上常规的全基因组随机测序的方法,把两个方法结合起来。
比如这有一堆的微生物,可能有1000个不同种类的微生物,采用经典的测序直接建库测序会得到一个基因组组装的结果,显然,这个组装的效果会非常差,也就意味着很多的信息都是片断化的、零碎的。
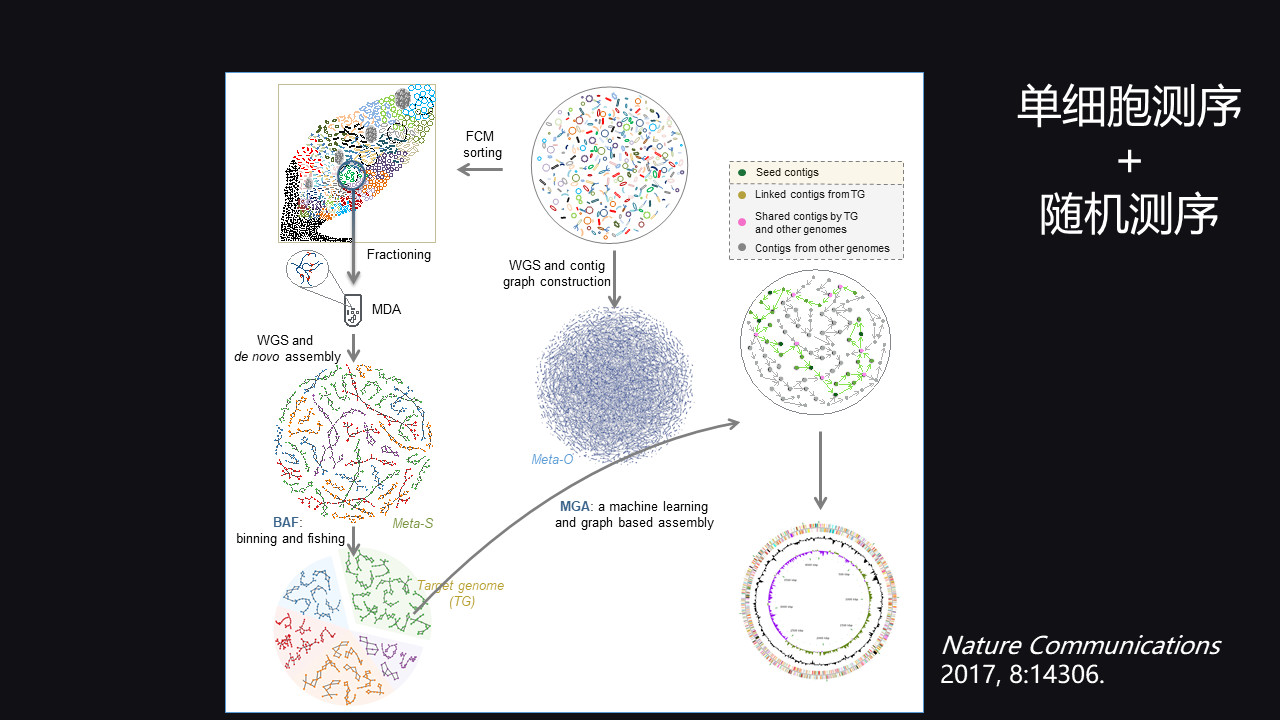
我们就想能不能把这些细胞、细菌分门别类,拆分成若干个子集,就像把整个江湖当中所有的人物,按门派来进行划分一样。
对于特定的子集,再去做后续的分选、建库、测序,就会得到一个小的 Meta(宏基因组),小的 Meta 的复杂度,要显著地低于原始的 Meta。
后续就可以采用相应的计算方法,把这个小的、低复杂度的和大的、高复杂度的结合起来,通过图论和机器学习的方法,就可以来获得相对高质量的微生物组的全序列。
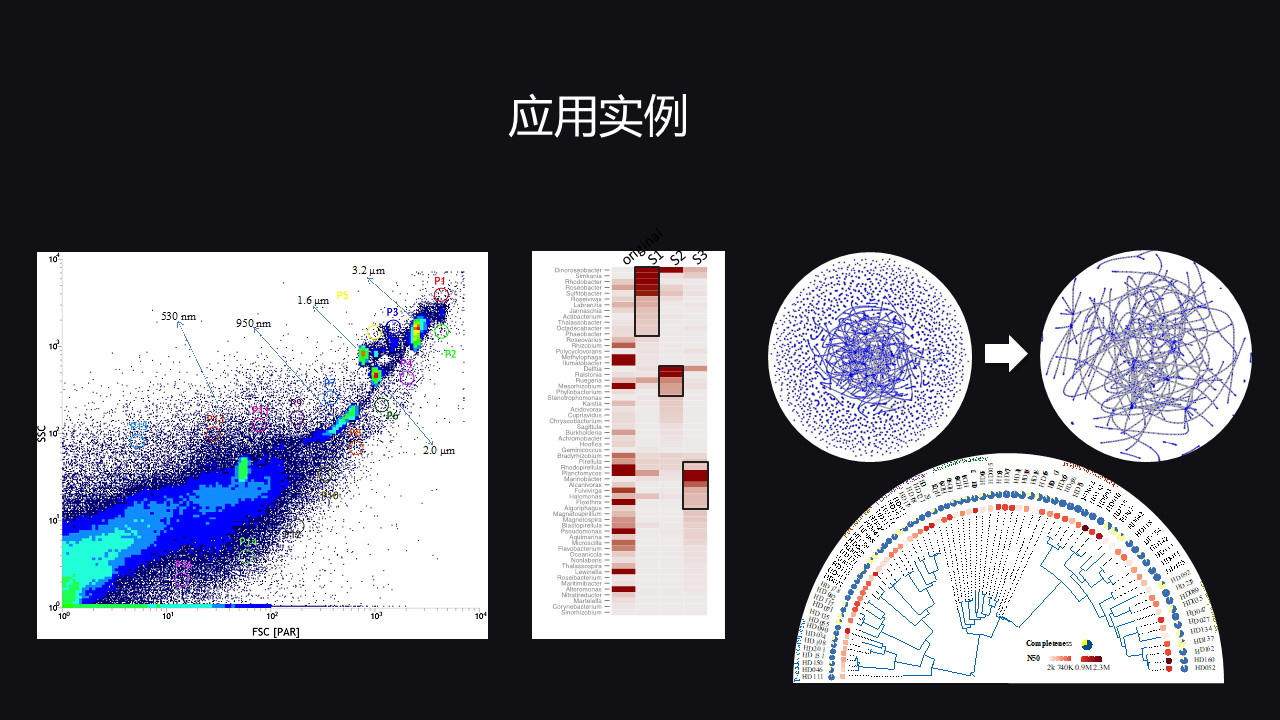
通过这个方法,可以看到它具有非常高的效率,因为可以通过控制分选数目的大小,去控制它的相应的数量。
当分选子集的数目非常非常少,细胞的数目非常少,少到只有一个,那就是单细胞测序。如果这个分选的数目非常非常大,就是原始的 Meta。

那可以看到,我们只分选了三个子集,就获得70多个接近完整的基因组,很显然这个数据,就有利于后面去分析它的功能基因,去分析这些细菌是如何适应环境,如何来影响人体健康的。
这些工作都是起点,最终我们仍然感兴趣的是这些细菌到底有什么用,它们在肠道当中是有益的、还是有害的?
这时候我们需要揭示这些细菌以及不同细菌之间,它们是怎么样相互作用,它们是互利共生,还是竞争拮抗,还是有着其他的什么样的关系?
这就类似于我们的江湖当中的,不同的门派他们的这种合纵连横。
对于菌群而言,它的关系的层次还是非常广泛,不仅仅有细菌之间的关系,而且有细菌和人体之间的关系。
很显然细菌可能会影响人体健康,人的遗传因素也会影响细菌的定殖。同样的我们的膳食营养会影响菌群的改变。
当然细菌也会有它自己的病毒,我们称之为噬菌体。噬菌体在菌群的平衡、丰度的维持上发挥着重要的作用。
那我们就可以采用相应的数据挖掘的方法,来去揭示这不同层次之间的相互关系,从而为解释我们菌群的作用打下基础。
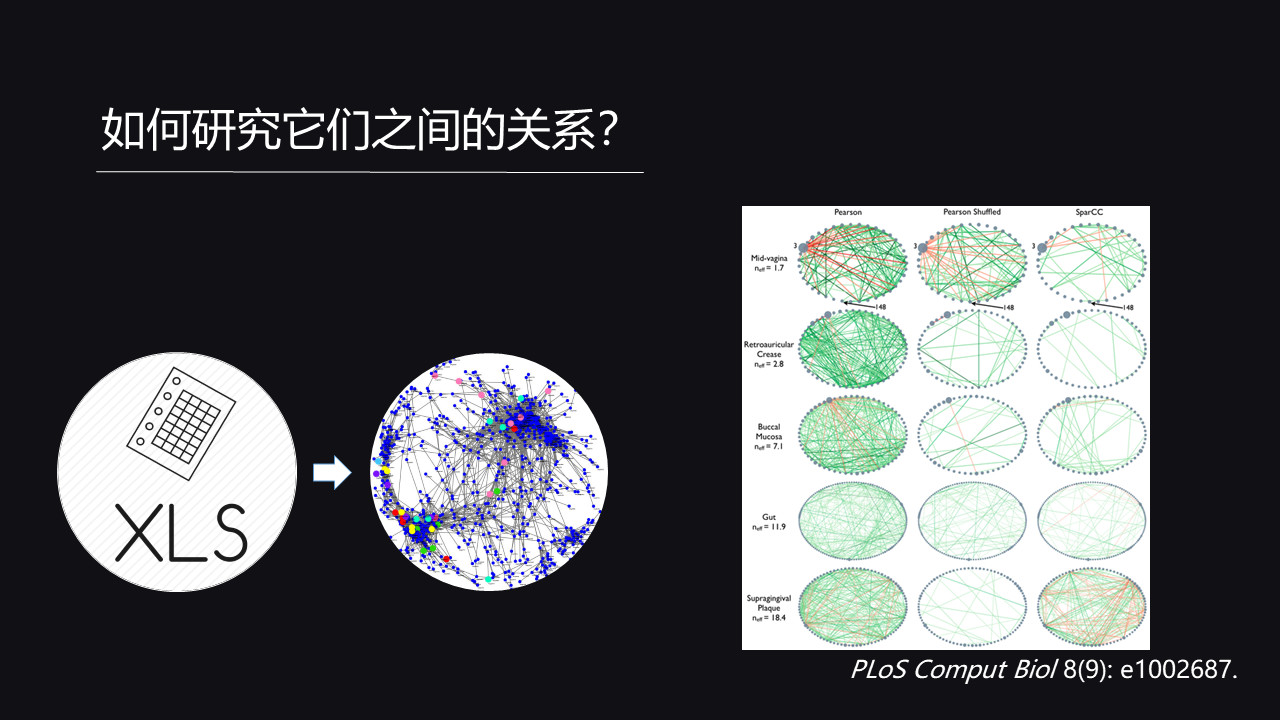
那么如何来研究它们之间的相互关系呢?那么这里面也会用到一些数据挖掘,或者说统计学模型的一些方法。
仍然举刚才的例子,咱们在座200多人,每一个人都有一个肠道的微生物的谱系,每个人里面有100种微生物,它的丰度彼此之间不一样。
那么我可以把这些所有的信息放到一个大的 EXCEL 表里面,每一行可以理解为某一个特定的一个细菌,每一列可以理解为在座的每一个人。
那么这表格里面的数字就意味着对于某一个特定菌,它在某一个特定人里面的生物量,就是它的丰度。
这个表格,可以用图形化的形式来表示出来,这个图形我们称之为一个交互的网络。
在这网络上,网络的节点就是意味着特定的两个菌之间的连线,就是它俩之间的相互关系。
这个连线可能是正相关,意味着两个菌它们是互利的,你多,我也多。如果这个相关是负相关,意味着你多,我就少。
通过这个网络就可以挖掘出来哪些细菌可能是共同发挥起作用,可能它会影响我们健康。
或者说如果我们想去掉一些特定的有害菌,那就去找哪些菌和它有竞争拮抗,我可以增加这些有益菌,即和其存在竞争拮抗的细菌,从而起到降低有害菌的作用。
当然在实际的数据挖掘的过程当中,它的问题的复杂性远远超过想象,里面有大量的噪音,有整个这个系数矩阵本身带来的一些计算的问题。
科学家们都可以采用种种的一些计算方法,一些统计学模型,来获得高质量的一些结果,来排除这其中的一些假阳性。
当重建了不同层次的菌群之间的关系的时候,就可以来利用这些关系帮助维护我们的健康。
这里面有一个大家所关注的,噬菌体治疗的这样一个提法,那这是什么意思?
因为噬菌体是可以侵染并且裂解它的宿主细菌,那么大家就想能不能利用噬菌体来替代抗生素?
大家知道,目前抗生素的耐药性可以说是全球普遍关注的一个问题,那么这里面有一个非常好的一个例子。
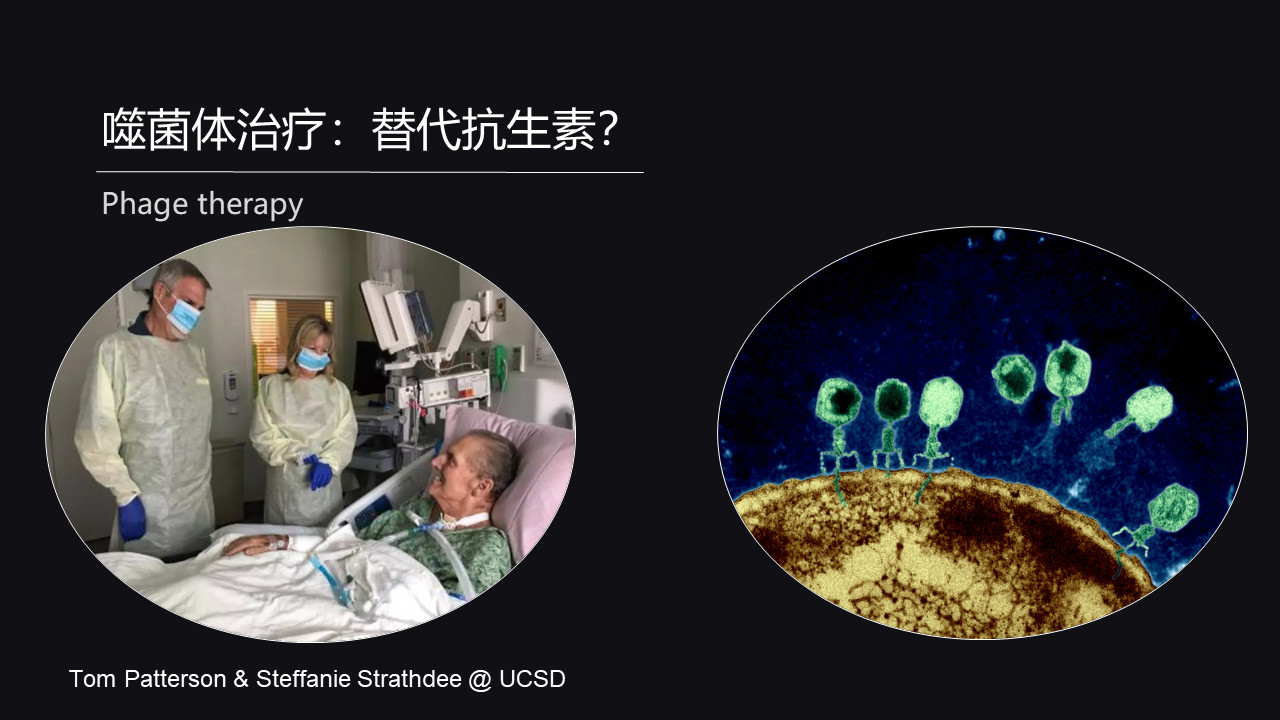
左边这个图显示的是美国的一个神经学家,他2015年去埃及旅游得了胰腺炎,辗转治疗从欧洲回到美国,治疗半年多越来越严重,最终临床检测,发现他感染了一种超级耐药的鲍曼不动杆菌。
他的夫人就是旁边站的这位女士也是加州 UCSD(加州大学圣地亚哥分校)的病原微生物学家。
她本身的专业知识让她知道,在抗生素无法治疗的时候可能需要一些其他的替代手段。
她就利用鲍曼不动杆菌的靶向的噬菌体来治疗,结果通过很短期的治疗就让她的先生恢复了健康。后面她也把相似的治疗方法用到更多的一些病例上。
这样的一个例子就充分告诉我们,这些基础研究的成果,完全可以用来改善或者治疗相应的一些疾病。
大家知道,抗生素具有广谱性,它不仅可以杀害一些有害菌,同时它也会误伤一些有益菌。
而噬菌体它具有高度的靶向性,它往往只能去侵染或者只能去针对某些特定种类的或者特定株系的细菌,很显然它就可以规避抗生素滥用的这样一些问题。
但总的来说,我们的想法就是如何能够从菌群数据当中获得真正有用的信息,来去帮助我们,去筛选这样的一些噬菌体。

常规的方法是借助于实验,比如我想去找某一个具体细菌的噬菌体的时候,可以设计各种各样的实验去捕获或者去富集相应的噬菌体,很显然这种方法低效、盲目、并且不可控。
我们就想能不能采用计算的方法。因为现在已经积累了海量的成千上万个人群的或者是个体的菌群的数据:包括肠道的、包括口腔的等等。这些数据里面就含有大量的数据的信息可供我们去挖掘、去研究。
这里面我们用到的一个方法就是这种 Cas9的 CRISPR 的结构。
当噬菌体侵染宿主细菌的时候,宿主细菌里面会有一套获得性免疫的一个机制,它可以把噬菌体的核酸给切碎,其中有一小段可能会被插入到 CRISPR 后面的这个 Spacer(间隔序列)的结构上面。
我们就想能不能识别这样一个串联重复的结构,基于结构信息就可以把噬菌体以及被它侵染的宿主的关系给找出来。
利用这个方法就可以建立这样一个网络,在这个网络当中,既有宿主噬菌体,也有相应的它对应的侵染细菌。
通过这个网络就可以帮助我们去进一步地挖掘它们之间可能存在什么样的一个侵染,这就为后续真正找到我们想要的具有潜在应用价值的噬菌体提供了一个数据的基础。

总的来说,菌群的高度复杂性不亚于人类社会,如何持续地挖掘和利用,如何把基础研究的成果转化为可临床应用的一些发现或者一些方法,我觉得我们仍然处于路上,仍然是任重而道远。
我相信这种基础研究的成果和临床的结合可以极大促进这一过程的转化。
好,谢谢大家。
(全文结束)


